渋谷駅前で働くデータサイエンティストのブログ
元祖「六本木で働くデータサイエンティスト」です / 道玄坂→銀座→東京→六本木→渋谷駅前
TensorFlow Probabilityを試してみる(1): 定番のEight SchoolsのモデリングをRStanと比較する
先日の記事でも書いたように、どうもここ最近RStan周りの環境が色々厳しくなっている気がしていて、仮にRStanが今後環境面での不具合やミスマッチなどで使えなくなったらベイジアンモデリングやれなくなって困るかも。。。という危惧を最近抱きつつあります。
そこで代替手段として思いつくのが、JAGS, PyMC, PyStan, そしてTensorFlow Probability (TFP)。TFPを挙げたのは完全に身贔屓なんですが(笑)、Pythonで回せるものとして近年注目を集めているフレームワークとしては筆頭に近いのではないかと思います。ということで、贔屓の引き倒しみたいになりそうですが今回含めてちょっと連続してTFPでRStanと同じことをやってみる、というただそれだけの備忘録的な記事をだらだらと書いていこうと思います。
いつもながらですが、僕はコーディングに関してはド素人ですので間違っている点・理解不足の点などあればどしどしご指摘ください。また、今回からPython 3.7で書いています。3系のコーディングについても誤りやおかしな点などあれば是非ご指摘いただければと思いますm(_ _)m
Eight Schools
今回は最初なので、RStanではそもそもインストール後の最初のテストとして使われる題材でもあるEight Schoolsを例に挙げます。これは先日僕もついに買ったGelmanの鈍器ことBDAの5.5節に出てくる階層ベイズモデル向けの実験データです。
簡単にデータの背景について述べておくと、8つの高校でUSの大学受験生向け標準テストであるSATのreadingパートであるSAT-Vの成績向上を目的として実施された特別な教育プログラムの効果を調べたものです。これは8つの高校それぞれで独立してランダム化比較試験が行われ、その教育プログラムがSAT-Vの点数(200〜800点の範囲で一般に平均500点&標準偏差100点とされる)をどれだけ向上させたかを調べており、データとしてその向上した点数とその標準偏差が記録されています(8行2列)。8つの高校で行われたプログラムは互いに同じであると仮定して良い、とします。
まず、RとPythonそれぞれでデータを与えます。
# For RStanschools_dat<-list(J=8, y= c(28,8,-3,7,-1,1,18,12), sigma= c(15,10,16,11,9,11,10,18))
# For TFPimport matplotlib.pyplotas pltimport numpyas npimport seabornas snsimport tensorflowas tfimport tensorflow_probabilityas tfpfrom tensorflow_probabilityimport distributionsas tfdimport warningsplt.style.use("ggplot")warnings.filterwarnings('ignore')# 8 schoolsnum_schools =8# number of schoolstreatment_effects = np.array( [28,8, -3,7, -1,1,18,12], dtype=np.float32)# treatment effectstreatment_stddevs = np.array( [15,10,16,11,9,11,10,18], dtype=np.float32)# treatment SEfig, ax = plt.subplots()plt.bar(range(num_schools), treatment_effects, yerr=treatment_stddevs)plt.title("8 Schools treatment effects")plt.xlabel("School")plt.ylabel("Treatment effect")fig.set_size_inches(10,8)plt.show()

見たまんまです。これに対してどういう階層ベイズモデルを与えるかというと、TFPのrepoに載っているモデルを採用するなら以下の通りになります。
ここでが教育プログラムの真の効果の平均、
がその学校ごとのばらつきにかかる係数、
が学校ごとにばらつく効果の大きさです。そして
と
は既に8行2列のデータとして手に入っている「学校ごとに測定された効果」と「その標準偏差」です。では、RStanとTFPとでそれぞれどうなるか見てみましょう。
RStanでやってみる
// saved as 8schools.standata {int<lower=0> J;// number of schools real y[J];// estimated treatment effects real<lower=0> sigma[J];// standard error of effect estimates}parameters { real mu;// population treatment effect real<lower=0> tau;// standard deviation in treatment effects vector[J] eta;// unscaled deviation from mu by school}transformed parameters { vector[J] theta = mu + tau * eta;// school treatment effects}model { target += normal_lpdf(eta |0,1);// prior log-density target += normal_lpdf(y | theta, sigma);// log-likelihood}
Getting Startedに載っているのでそのまま回すだけです(笑)。ただしTFPのrepoに書かれたモデル式と若干変数名が違うので、そこは適当に置き換えて読んでください。
library(rstan)options(mc.cores= parallel::detectCores())rstan_options(auto_write=TRUE)fit<- stan(file='8schools.stan', data= schools_dat, iter=1000, chains=4)plot(fit, pars= c('mu','tau','eta'))plot(fit, pars='theta')

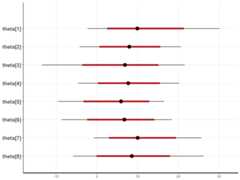
教育プログラム全体の平均的効果であるが意外とばらつきが大きいことが分かります。これに対して8つの学校ごとの「個体差」を示す
は互いに重なり合う範囲に分布しており、学校ごとではそれほど大きな差を示していません。
library(bayesplot)mcmc_sample<- extract(fit, permuted=F)mcmc_combo(mcmc_sample, pars= c('eta[1]','eta[2]','eta[3]','eta[4]'))mcmc_combo(mcmc_sample, pars= c('eta[5]','eta[6]','eta[7]','eta[8]'))


extract関数でMCMCサンプルを抽出してきてから{bayesplot}を使えば、chainと分布をプロットして収束度合いを確認することもできます。
TFPでやってみる
そもそもTFPのrepoにnotebookが載っています(笑)。なので単にそれをなぞるだけですが、一応RStanとの差異を見ておきましょう。
model = tfd.JointDistributionSequential([ tfd.Normal(loc=0., scale=10., name="avg_effect"),# `mu` above tfd.Normal(loc=5., scale=1., name="avg_stddev"),# `log(tau)` above tfd.Independent(tfd.Normal(loc=tf.zeros(num_schools), scale=tf.ones(num_schools), name="school_effects_standard"),# `theta_prime` reinterpreted_batch_ndims=1),lambda school_effects_standard, avg_stddev, avg_effect: ( tfd.Independent(tfd.Normal(loc=(avg_effect[..., tf.newaxis] + tf.exp(avg_stddev[..., tf.newaxis]) * school_effects_standard),# `theta` above scale=treatment_stddevs), name="treatment_effects",# `y` above reinterpreted_batch_ndims=1))])deftarget_log_prob_fn(avg_effect, avg_stddev, school_effects_standard):"""Unnormalized target density as a function of states."""return model.log_prob(( avg_effect, avg_stddev, school_effects_standard, treatment_effects))
ここがモデル部分ですが、オリジナルのTF的な要素とPython的な要素が入り混じって、RStanに慣れた身からするとちょっと取っ付きにくい感じがあります(汗)。
特徴的なのはtfd.JointDistributionSequentialです。これはAPIのドキュメントを見た方が分かりやすいかもしれません。
tfd = tfp.distributions# Consider the following generative model:# e ~ Exponential(rate=[100,120])# g ~ Gamma(concentration=e[0], rate=e[1])# n ~ Normal(loc=0, scale=2.)# m ~ Normal(loc=n, scale=g)# for i = 1, ..., 12:# x[i] ~ Bernoulli(logits=m)# In TFP, we can write this as:joint = tfd.JointDistributionSequential([ tfd.Independent(tfd.Exponential(rate=[100,120]),1),# elambda e: tfd.Gamma(concentration=e[...,0], rate=e[...,1]),# g tfd.Normal(loc=0, scale=2.),# nlambda n, g: tfd.Normal(loc=n, scale=g)# mlambda m: tfd.Sample(tfd.Bernoulli(logits=m),12)# x])# (Notice the 1:1 correspondence between "math" and "code".)
個人的な感覚としては、TFでtensorを流すためのgraphを書くのと同じノリなのかなと。ベイジアン的な生成モデルのgraphを、Stanで書くのとはまた異なる流儀で書いていく感じです。順番に確率分布を積み重ねていくと、最終的な尤度関数が得られるので、これをMCMCでパラメータ推定する*1ということですね。
あと、肝になるのがtfd.Independentなんですが公式ドキュメントを見てもいまいち良く分からなかったので、こちらの記事を参照しました。
元の確率分布のshapeを狙った形のshapeに変える(切り出す)ためのメソッド、という理解で良いのでしょうか? またPythonに不慣れな僕にはさらに不慣れなlambda式なども入り混じっていてゴツい印象を受けますが、ともあれこれらをsequentialに組み合わせることで生成モデルを表現できるようだということは分かりました。
num_results =5000num_burnin_steps =3000# Improve performance by tracing the sampler using `tf.function`# and compiling it using XLA.@tf.function(autograph=False, experimental_compile=True)defdo_sampling():return tfp.mcmc.sample_chain( num_results=num_results, num_burnin_steps=num_burnin_steps, current_state=[ tf.zeros([], name='init_avg_effect'), tf.zeros([], name='init_avg_stddev'), tf.ones([num_schools], name='init_school_effects_standard'), ], kernel=tfp.mcmc.HamiltonianMonteCarlo( target_log_prob_fn=target_log_prob_fn, step_size=0.4, num_leapfrog_steps=3))states, kernel_results = do_sampling()avg_effect, avg_stddev, school_effects_standard = statesschool_effects_samples = ( avg_effect[:, np.newaxis] + np.exp(avg_stddev)[:, np.newaxis] * school_effects_standard)num_accepted = np.sum(kernel_results.is_accepted)print('Acceptance rate: {}'.format(num_accepted / num_results))
実際のMCMC*2サンプリング部分。RStanとサンプリング部分にかかる時間は同じかちょっと早いぐらいかもですが、コンパイルの時間がかからない分体感としてはかなり早く感じられます。ちなみにオリジナルのnotebookではacceptance rateは0.5974でしたが、僕の手元でやった時は0.6002でした。
fig, axes = plt.subplots(8,2, sharex='col', sharey='col')fig.set_size_inches(12,10)for iinrange(num_schools): axes[i][0].plot(school_effects_samples[:,i].numpy()) axes[i][0].title.set_text("School {} treatment effect chain".format(i)) sns.kdeplot(school_effects_samples[:,i].numpy(), ax=axes[i][1], shade=True) axes[i][1].title.set_text("School {} treatment effect distribution".format(i))axes[num_schools -1][0].set_xlabel("Iteration")axes[num_schools -1][1].set_xlabel("School effect")fig.tight_layout()plt.show()

8つの学校ごとの教育プログラムの「個体差」のMCMCサンプルのchainとその分布を図示するコードです。この辺はRStanにおける{bayesplot}的な親切なパッケージやメソッドがあっても良いかなぁと思います。
# Compute the 95% interval for school_effectsschool_effects_low = np.array([ np.percentile(school_effects_samples[:, i],2.5)for iinrange(num_schools)])school_effects_med = np.array([ np.percentile(school_effects_samples[:, i],50)for iinrange(num_schools)])school_effects_hi = np.array([ np.percentile(school_effects_samples[:, i],97.5)for iinrange(num_schools)])fig, ax = plt.subplots(nrows=1, ncols=1, sharex=True)ax.scatter(np.array(range(num_schools)), school_effects_med, color='red', s=60)ax.scatter( np.array(range(num_schools)) +0.1, treatment_effects, color='blue', s=60)plt.plot([-0.2,7.4], [np.mean(avg_effect), np.mean(avg_effect)],'k', linestyle='--')ax.errorbar( np.array(range(8)), school_effects_med, yerr=[ school_effects_med - school_effects_low, school_effects_hi - school_effects_med ], fmt='none')ax.legend(('avg_effect','HMC','Observed effect'), fontsize=14)plt.xlabel('School')plt.ylabel('Treatment effect')plt.title('HMC estimated school treatment effects vs. observed data')fig.set_size_inches(10,8)plt.show()

MCMCから推定した教育プログラムの効果の推定値と、データから読み取れる実測値&ばらつきと、平均効果とを8つの学校それぞれについてまとめてプロットしたものです。大体RStanによる結果と同じように見えます。
 id:TJO
id:TJOTakashi J. OZAKI, Ph.D.
Data Scientist (尾崎 隆)
English:https://tjo-en.hatenablog.com/
このブログにはApache 2.0ライセンスのもとで配布されている製作物が含まれています。
ブログの内容は個人の意見・見解の表明であり、所属組織の意見・見解を代表しません。またブログ内容の正確性については一切保証いたしません(誤りを見つけた場合はコメント欄などでお知らせいただけると有難いです)。
また、ブログの中で取り上げられているデータ分析事例・データセット・分析上の知見など全ての記述は、特に明記されていない限りは、いずれもいかなる実在する企業・組織・機関の、いかなる個別の事例とも無関係です。ブログ記事内容は予告なく公開後に改変されることがあります。改変した事実は明示されることもあれば明示されないこともあります。
現在、講演依頼・書籍執筆依頼・メディア取材及び出演依頼等は全てお断りしております。悪しからずご了承ください。
- プロフィール:LinkedIn
- ご質問など:Quora
- 業績一覧:Google Scholar Citations
- 科研費情報:KAKEN
ご連絡はLinkedInメッセージでお願いいたします。
Copyright © Takashi J. OZAKI 2013 All rights reserved.
- AI・人工知能 (14)
- 論文 (21)
- 生成AI (7)
- 機械学習 (188)
- 統計学 (174)
- ベイズ統計学 (5)
- Python (43)
- MMM (3)
- データサイエンス (46)
- データ分析 (106)
- 統計的因果推論 (27)
- ビジネス (110)
- マーケティング (30)
- 再現性問題 (7)
- 時系列分析 (54)
- 旅行記 (19)
- R (162)
- LLM (9)
- 雑感 (31)
- エイプリルフール (12)
- 書籍 (55)
- 書評 (45)
- Deep Learning (33)
- 年末回顧 (9)
- BUGS/Stan (26)
- 人材 (59)
- LLMで試すシリーズ (2)
- SQL (2)
- graph/network (9)
- データサイエンティスト (109)
- TensorFlow (17)
- 研究 (4)
- キャリア (18)
- サンプルデータで試す機械学習シリーズ (19)
- MCMC (14)
- 回顧録 (10)
- DeepLearning実践シリーズ (11)
- 私事 (8)
- 機械学習エンジニア (29)
- 生TensowFlow七転八倒記 (13)
- 最適化計画 (3)
- データ分析実践編 (5)
- 機械学習の自動化 (6)
- DLM (9)
- AutoML (4)
- 状態空間 (2)
- 自己紹介 (3)
- 異常検知 (4)
- お知らせ (8)
- PR (4)
- Matlab (2)
- カンファレンス (8)
- テキストマイニング (1)
- 日常 (3)
- アナリティクス (10)
- Hadoop (2)
- Hive (2)
- Excel (1)
- Facebook (2)
- IT (1)

