KR20180128864A - Gene editing composition comprising sgRNAs with matched 5' nucleotide and gene editing method using the same - Google Patents
Gene editing composition comprising sgRNAs with matched 5' nucleotide and gene editing method using the sameDownload PDFInfo
- Publication number
- KR20180128864A KR20180128864AKR1020180059165AKR20180059165AKR20180128864AKR 20180128864 AKR20180128864 AKR 20180128864AKR 1020180059165 AKR1020180059165 AKR 1020180059165AKR 20180059165 AKR20180059165 AKR 20180059165AKR 20180128864 AKR20180128864 AKR 20180128864A
- Authority
- KR
- South Korea
- Prior art keywords
- cas9
- lys
- leu
- sequence
- sgrna
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Granted
Links
Images
Classifications
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/10—Processes for the isolation, preparation or purification of DNA or RNA
- C12N15/102—Mutagenizing nucleic acids
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/113—Non-coding nucleic acids modulating the expression of genes, e.g. antisense oligonucleotides; Antisense DNA or RNA; Triplex- forming oligonucleotides; Catalytic nucleic acids, e.g. ribozymes; Nucleic acids used in co-suppression or gene silencing
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/87—Introduction of foreign genetic material using processes not otherwise provided for, e.g. co-transformation
- C12N15/90—Stable introduction of foreign DNA into chromosome
- C12N15/902—Stable introduction of foreign DNA into chromosome using homologous recombination
- C12N15/907—Stable introduction of foreign DNA into chromosome using homologous recombination in mammalian cells
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/14—Hydrolases (3)
- C12N9/16—Hydrolases (3) acting on ester bonds (3.1)
- C12N9/22—Ribonucleases [RNase]; Deoxyribonucleases [DNase]
Landscapes
- Life Sciences & Earth Sciences (AREA)
- Health & Medical Sciences (AREA)
- Genetics & Genomics (AREA)
- Engineering & Computer Science (AREA)
- Chemical & Material Sciences (AREA)
- Biomedical Technology (AREA)
- Zoology (AREA)
- Organic Chemistry (AREA)
- Bioinformatics & Cheminformatics (AREA)
- Wood Science & Technology (AREA)
- Molecular Biology (AREA)
- Biotechnology (AREA)
- General Engineering & Computer Science (AREA)
- General Health & Medical Sciences (AREA)
- Biochemistry (AREA)
- Microbiology (AREA)
- Physics & Mathematics (AREA)
- Plant Pathology (AREA)
- Biophysics (AREA)
- Mycology (AREA)
- Cell Biology (AREA)
- Crystallography & Structural Chemistry (AREA)
- Medicinal Chemistry (AREA)
- Micro-Organisms Or Cultivation Processes Thereof (AREA)
- Enzymes And Modification Thereof (AREA)
Abstract
Translated fromKoreanDescription
Translated fromKorean매칭된 5' 뉴클레오타이드를 포함하는 가이드 RNA를 이용하여 고특이성 Cas9 변이체의 유전자 교정 효율을 증진시키는 기술과 관련된 것으로, 고특이성 Cas9 변이체 및 매칭된 5' 뉴클레오타이드를 포함하는 가이드 RNA의 복합체, 고특이성 Cas9 변이체 및 매칭된 5' 뉴클레오타이드를 포함하는 유전자 교정용 조성물; 및 유전자 교정용 조성물을 이용하는 유전자 교정 방법이 제공된다.The present invention relates to a technique for enhancing the gene correction efficiency of a high specificity Cas9 mutant using a guide RNA comprising a matched 5 'nucleotide, comprising a complex of guide RNA comprising a high specificity Cas9 mutant and a matched 5' nucleotide, a high specificity Cas9 A mutant and a matched 5 'nucleotide; And a gene correction method using a composition for gene correction are provided.
박테리아와 고세균 내의 적응 면역 시스템 (adaptive immune system)에서 유래한 CRISPR-Cas9 RNA-가이드 엔도뉴클레아제는 다양한 세포 및 유기체 내에서의 표적화된 유전체 교정 용도로 용도 변경되었다. 이들 CRISPR-Cas9 RNA-가이드 엔도뉴클레아제는 염색체 DNA를 표적화된 방식으로 절단하여 부위특이적 DNA 이중 가닥 절단 (DSB; double strand break)을 생성하며, 비상동성 말단 결합 (NHEJ; non-homologous end-joining)을 통한 repair는 표적 부위에서 삽입(insertion) 또는 결실(deletion) (indels)을 유도한다. 불행히도, 표적 부위와 서열 상동성이 높은 부위에서의 표적 DNA 절단은 원하지 않는 유전체 자리에서의 돌연변이 및 염색체 재배열을 유도할 수 있다 (off-target effect).S.pyogenes Cas9 뉴클레아제 및 가이드 RNA (sgRNA) 모두 이러한 off-target effect를 최소화하거나 제거하기 위한 변형이 가해져 왔다. 특히, 인간 세포에서 최소화 내지는 탐지되지 않는 정도로 낮은 off-target effect를 갖는 고특이성 Cas9 변이체가 개발되었으며, 그 예로 enhanced Cas9-1.1 (eCas9-1.1)(Slaymaker, I.M. et al. Rationally engineered Cas9 nucleases with improved specificity.Science351, 84-88 (2016)) 및 Cas9 high-fidelity variant 1 (Cas9-HF1) (Kleinstiver, B.P. et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects.Nature529, 490-495 (2016))가 있다. 이들 고특이성 Cas9 변이체들은 알라닌 치환을 포함하여 Cas9 단백질과 비-표적 또는 표적 DNA 가닥 사이의 비특이적인 이온 상호작용을 약화시킨다.The CRISPR-Cas9 RNA-guided endonuclease, which is derived from an adaptive immune system in bacteria and archaea, has been modified for targeted genetic modification applications in a variety of cells and organisms. These CRISPR-Cas9 RNA-guided endonucleases cleave chromosomal DNA in a targeted fashion to produce site-specific double strand breaks (DSBs), and non-homologous end (NHEJ) -joining leads to insertion or deletion (indels) at the target site. Unfortunately, target DNA cleavage at sites that are highly homologous to the target site can lead to mutations and chromosomal rearrangements at the undesired genomic site (off-target effect).S.Both pyogenes Cas9 nuclease and guide RNA (sgRNA) have been modified to minimize or eliminate this off-target effect. In particular, highly specific Cas9 variants with minimal or undetectable off-target effect in human cells have been developed, such as enhanced Cas9-1.1 (eCas9-1.1) (Slaymaker, IM et al., Rationally engineered Cas9 nucleases with improvedspecificity. Science 351, 84-88 (2016 )) and Cas9 high-fidelity variant 1 (Cas9 -HF1) (Kleinstiver, BP et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature529 , 490-495 (2016)). These high specificity Cas9 mutants, including alanine substitutions, weaken nonspecific ionic interactions between Cas9 protein and non-target or target DNA strands.
이와 같은 고특이성 Cas9 변이체들의 유전자 교정 활성 및 특이성을 보다 증진시키기 위한 기술의 개발이 요구된다.There is a need to develop a technique for further enhancing the gene correcting activity and specificity of such high specificity Cas9 variants.
본 명세서는 고특이성 Cas9 변이체들의 오프-타겟 부위에 대한 낮은 유전자 교정 활성을 유지하면서 온-타겟 특이적 유전자 교정 활성을 보다 증진시킬 수 있는 기술을 제공한다. 보다 구체적으로, 매칭된 5' 뉴클레오타이드를 포함하는 가이드 RNA (예컨대, sgRNA)를 사용하여 Cas9 변이체의 유전자 교정 효율 (예컨대, 인델 (indel) 빈도)을 증가시키는 기술을 제공한다.This specification provides a technique that can further enhance on-target specific gene correcting activity while maintaining low genetic corrective activity against off-target regions of high specificity Cas9 variants. More specifically, there is provided a technique for increasing the gene correction efficiency (e.g., indel frequency) of a Cas9 mutant using a guide RNA (e.g., sgRNA) comprising a matched 5 'nucleotide.
일 예는 고특이성 Cas9 변이체 및 매칭된 5' 뉴클레오타이드를 포함하는 가이드 RNA(예컨대, sgRNA)의 복합체를 제공한다.One example provides a complex of guide RNA (e.g., sgRNA) comprising a high specificity Cas9 mutant and a matched 5 'nucleotide.
다른 예는 고특이성 Cas9 변이체 및 매칭된 5' 뉴클레오타이드를 포함하는 가이드 RNA(예컨대, sgRNA)를 포함하는 유전자 교정용 조성물을 제공한다.Another example provides a composition for gene correction comprising a guide RNA (e.g., sgRNA) comprising a high specificity Cas9 variant and a matched 5 'nucleotide.
다른 예는 고특이성 Cas9 변이체 및 매칭된 5' 뉴클레오타이드를 포함하는 가이드 RNA(예컨대, sgRNA)를 사용하는 유전자 교정 방법을 제공한다. 예컨대, 상기 방법은 상기 고특이성 Cas9 변이체 및 매칭된 5' 뉴클레오타이드를 포함하는 가이드 RNA(예컨대, sgRNA)를 표적 유전자 또는 상기 표적 유전자 내에 위치하는 표적 부위 (PAM 서열을 포함하는, 10 내지 30개 뉴클레오타이드, 10 또는 25개 뉴클레오타이드, 15 내지 30개 뉴클레오타이드, 15 또는 25개 뉴클레오타이드, 17 내지 30개 뉴클레오타이드, 또는 17 또는 25개 뉴클레오타이드, 예컨대 20개 뉴클레오타이드)와 접촉시키는 단계를 포함할 수 있으며, 상기 접촉시키는 단계는 상기 복합체를 대상에 투여, 주입, 또는 도입함으로써 수행되는 것일 수 있다.Another example provides a method of gene correction using a guide RNA (e.g., sgRNA) comprising a high specificity Cas9 mutant and a matched 5 'nucleotide. For example, the method may comprise introducing a guide RNA (e.g., sgRNA) comprising the high specificity Cas9 mutant and a matched 5 'nucleotide into a target gene or a target site (PAM sequence containing 10 to 30 nucleotides , 10 or 25 nucleotides, 15-30 nucleotides, 15 or 25 nucleotides, 17-30 nucleotides, or 17 or 25 nucleotides such as 20 nucleotides), and the contacting The step may be performed by administering, injecting, or introducing the complex into a subject.
상기 고특이성 Cas9 변이체는 하나 이상의 아미노산이 알라닌으로 치환되어 표적 부위에 대한 특이성이 증진된 Cas9 변이체를 의미하는 것으로, 예컨대,Streptococcuspyogenes Cas9 단백질의 아미노산 서열 (서열번호 4)을 기준으로, K848, K1003, R1060, N497, R661, Q695, 및 Q926로 이루어진 군에서 선택된 하나 이상의 아미노산이 알라닌으로 치환된 Cas9 변이체를 의미하는 것일 수 있다. 일 구체예에서, 상기 고특이성 Cas9 변이체는Streptococcuspyogenes Cas9 단백질(서열번호 4)에 K848A, K1003A, 및 R1060A 변이가 도입된 eCas9-1.1, 또는 N497A, R661A, Q695A, 및 Q926A 변이가 도입된 Cas9-HF1, 또는 이들의 조합일 수 있다.The high specificity Cas9 mutant means a Cas9 variant in which at least one amino acid has been replaced with alanine to increase specificity to a target site. For example, K848, K1003 (SEQ ID NO: 4) of theStreptococcuspyogenes Cas9 protein , R1060, N497, R661, Q695, and Q926 may be substituted with alanine. In one embodiment, the high specificity Cas9 mutant is Cas9- 1. 1 in which the K848A, K1003A, and R1060A mutations are introduced into theStreptococcuspyogenes Cas9 protein (SEQ ID NO: 4), or Cas9-1.1 in which the N497A, R661A, Q695A, and Q926A mutations are introduced. HF1, or a combination thereof.
본 명세서에서, 가이드 RNA서 매칭된 5' 뉴클레오타이드는 각 가이드 RNA가 표적하는 표적 서열의 가장 5' 말단에 위치하는 뉴클레오타이드 (5' 뉴클레오타이드라 칭함)와 매칭되는 (일치하는) 염기를 포함하는 뉴클레오타이드를 의미한다.Herein, the 5 'nucleotide matched with the guide RNA includes a nucleotide containing a base matched with (coincident with) a nucleotide (referred to as 5' nucleotide) located at the 5 'end of the target sequence targeted by each guide RNA it means.
다른 예는 상기 유전자 교정 방법에 의하여 교정된 유전자를 포함하는 유전자 변형 세포를 제공한다.Another example provides a genetically modified cell comprising the gene corrected by the above gene correction method.
다른 예는 상기 유전자 변형 세포로부터 얻어진 유전자 변형 동물을 제공한다.Another example provides a transgenic animal obtained from said transgenic cell.
상기 방법, 복합체 및 조성물은 진핵 세포 (예컨대, 분리된 인간 세포, 또는 인간을 제외한 진핵 동물 또는 진핵 식물의 세포) 또는 진핵 유기체 (예컨대, 인간, 또는 인간을 제외한 인간을 제외한 진핵 동물 또는 진핵 식물)에 적용되는 것일 수 있다.The methods, complexes, and compositions can be used to treat eukaryotic cells (e. G., Isolated human cells, or eukaryotic or eukaryotic cells other than humans) or eukaryotic organisms (e. G., Human, As shown in FIG.
다른 예는 (1) 가이드 RNA (예컨대, sgRNA) 및 (2) 상기 가이드 RNA의 5' 말단에 융합된 자가-절단 활성을 갖는 RNA 절단효소 또는 1 내지 6개의 tRNA를 포함하는, 융합 RNA 분자를 제공한다.Another example is a fusion RNA molecule comprising (1) a guide RNA (e. G., SgRNA) and (2) an RNA cleavage enzyme having a self-cleaving activity fused to the 5 ' to provide.
다른 예는 가이드 RNA (예컨대, sgRNA)의 5' 말단, 3' 말단, 또는 양 말단에 자가-절단 활성을 갖는 RNA 절단효소 또는 1 내지 6개의 tRNA를 융합시키는 단계를 포함하는, 표적 서열과 매칭된 5' 말단 뉴클레오타이드를 포함하는 가이드 RNA의 제조 방법을 제공한다. 상기 제조 방법에서 융합시키는 단계는 가이드 RNA (예컨대, sgRNA)의 암호화 DNA와 자가-절단 활성을 갖는 RNA 절단효소 또는 1 내지 6개의 tRNA를 암호화하는 DNA를 하나의 벡터에서 발현시키는 단계를 포함할 수 있다.Another example is to match a target sequence comprising a step of fusing an RNA cleaving enzyme or one to six tRNAs having a self-cleaving activity at the 5'-, 3'-, or both ends of a guide RNA (e.g., sgRNA) Lt; / RTI > nucleotides and a 5 ' -terminal nucleotide. The step of fusing in the above production method may include the step of expressing in a vector a DNA coding for an encoding DNA of a guide RNA (for example, sgRNA) and an RNA cleaving enzyme having self-cleavage activity or 1 to 6 tRNA have.
상기 자가-절단 활성을 갖는 RNA 절단효소(리보자임)은 해머해드 리보자임 (hammerhead ribozyme; 예컨대, Type I hammerhead ribozyme, Type II hammerhead ribozyme, TypeIII hammerhead ribozyme 등), VS (Varkud satellite) 리보자임, 리드자임 (Leadzyme), 헤어핀 리보자임 (hairpin ribozyme) 등으로 이루어진 군에서 선택된 1종 이상일 수 있으나, 이에 제한되는 것은 아니다.(Ribozyme) having the self-cleaving activity may be selected from the group consisting of hammerhead ribozyme (for example, type I hammerhead ribozyme, type II hammerhead ribozyme, type III hammerhead ribozyme and the like), VS (Varkud satellite) ribozyme, A hairpin ribozyme, and the like, but the present invention is not limited thereto.
진핵 세포에서 sgRNA를 발현하는데 일반적으로 사용되는 U6 프로모터는 전사를 개시하기 위하여 구아노신(G) 뉴클레오타이드를 필요로 하기 때문에 sgRNA는 전형적으로 5' 말단에 "G" 뉴클레오타이드를 포함한다. 대부분의 (평균 75%) DNA 표적 부위는 이 위치에서 미스매치 뉴클레오타이드 (즉 G가 아닌 염기 (A, T, 또는 C)를 포함하는 뉴클레오타이드)를 포함한다. 본 발명자들은 고특이성 Cas9 변이체를 5' 말단에 G를 포함하는 가이드 RNA와 함께 5' 말단에 G를 포함하지 않는 표적 서열에 사용하는 경우, 5' 말단에 가이드 RNA와 표적 서열 간 미스매치가 발생하여, 상기 표적 서열 부위에서의 상기 고특이성 Cas9 변이체에 의한 유전자 교정 효율이 낮아짐을 최초로 확인하여, 고특이성 Cas9 변이체의 유전자 교정 효율에 있어서 가이드 RNA의 5' 말단과 표적 서열의 5' 말단의 매칭 여부가 중요한 역할을 가짐을 제안한다.Because the U6 promoter commonly used to express sgRNA in eukaryotic cells requires a guanosine (G) nucleotide to initiate transcription, sgRNA typically contains a " G " nucleotide at the 5 ' end. Most (average 75%) DNA target sites contain mismatch nucleotides (ie, nucleotides containing a base (A, T, or C) other than G) at this position. The present inventors have found that when a high specificity Cas9 mutant is used in a target sequence not including G at the 5 'end together with a guide RNA containing G at the 5' end, a mismatch between the guide RNA and the target sequence occurs at the 5 ' , It was confirmed that the efficiency of gene correction by the high specificity Cas9 mutant at the target sequence region was lowered. As a result, in the gene correction efficiency of the high specificity Cas9 mutant, the 5 'end of the guide RNA and the 5' Suggesting that it has an important role to play.
또한, 자기-절단 리보자임 (self-cleaving ribozyme)에 연결되어 생산된 매칭된 5' 뉴클레오타이드를 포함하는 sgRNA를 사용함으로써, 인간을 포함한 진핵 세포에서, 상기 고특이성 Cas9 변이체가 오프-타겟 부위에 낮은 활성을 보이는 고특이성(high-specificity)을 희생시키지 않으면서 온-타겟 부위에서의 유전자 교정 활성을 현저하게 향상시킬 수 있음을 확인하였다.Also, by using sgRNAs comprising matched 5 'nucleotides produced in connection with self-cleaving ribozymes, it has been shown that, in eukaryotic cells, including humans, the high specificity Cas9 mutant has a low It was confirmed that the gene correcting activity at the on-target site can be remarkably improved without sacrificing high-specificity showing activity.
이에, 본 발명의 일 예는 고특이성 Cas9 변이체 및 매칭된 5' 뉴클레오타이드를 포함하는 가이드 RNA(예컨대, sgRNA)의 복합체를 제공한다.Thus, an example of the present invention provides a complex of guide RNA (e.g., sgRNA) comprising a high specificity Cas9 mutant and a matched 5 'nucleotide.
다른 예는 고특이성 Cas9 변이체 및 매칭된 5' 뉴클레오타이드를 포함하는 가이드 RNA(예컨대, sgRNA)를 포함하는 유전자 교정용 조성물을 제공한다.Another example provides a composition for gene correction comprising a guide RNA (e.g., sgRNA) comprising a high specificity Cas9 variant and a matched 5 'nucleotide.
다른 예는 고특이성 Cas9 변이체 및 매칭된 5' 뉴클레오타이드를 포함하는 가이드 RNA(예컨대, sgRNA)를 사용하는 유전자 교정 방법을 제공한다. 예컨대, 상기 방법은 고특이성 Cas9 변이체 및 매칭된 5' 뉴클레오타이드를 포함하는 가이드 RNA(예컨대, sgRNA)를 표적 유전자 또는 상기 표적 유전자 내에 위치하는 표적 부위 (PAM 서열을 포함하는, 13 내지 30개 뉴클레오타이드, 13 또는 25개 뉴클레오타이드, 15 내지 30개 뉴클레오타이드, 15 또는 25개 뉴클레오타이드, 20 내지 30개 뉴클레오타이드, 또는 20 또는 25개 뉴클레오타이드)와 접촉시키는 단계를 포함할 수 있으며, 상기 접촉시키는 단계는 상기 복합체를 대상 (진핵 세포 또는 진핵 유기체)에 투여, 주입, 또는 도입함으로써 수행되는 것일 수 있다.Another example provides a method of gene correction using a guide RNA (e.g., sgRNA) comprising a high specificity Cas9 mutant and a matched 5 'nucleotide. For example, the method may comprise introducing a guide RNA (e.g., sgRNA) comprising a high specificity Cas9 mutant and a matched 5 'nucleotide into a target gene or a target site located within the target gene (13 to 30 nucleotides, 13 or 25 nucleotides, 15-30 nucleotides, 15 or 25 nucleotides, 20-30 nucleotides, or 20 or 25 nucleotides), said contacting comprising contacting said complex with a target (Eukaryotic cell or eukaryotic organism).
상기 고특이성 Cas9 변이체는 알라닌이 아닌 하나 이상의 아미노산 잔기가 알라닌으로 치환되어 표적 부위에 대한 특이성이 증진된 Cas9 변이체를 의미하는 것으로, 본 명세서에서 고충실도 (high-fidelity) Cas9 변이체라고도 명명된다. 예컨대, 상기 고특이성 Cas9 변이체는Streptococcuspyogenes Cas9 단백질의 아미노산 서열 (서열번호 4)을 기준으로, K848, K1003, R1060, N497, R661, Q695, 및 Q926로 이루어진 군에서 선택된 하나 이상의 아미노산이 알라닌으로 치환된 Cas9 변이체를 의미하는 것일 수 있다. 일 구체예에서, 상기 고특이성 Cas9 변이체는Streptococcuspyogenes Cas9 단백질(서열번호 4)에 K848A, K1003A, 및 R1060A 변이가 도입된 eCas9-1.1, 또는 N497A, R661A, Q695A, 및 Q926A 변이가 도입된 Cas9-HF1, 또는 이들의 조합일 수 있다.The high specificity Cas9 mutant refers to a Cas9 mutant in which at least one amino acid residue that is not alanine is substituted with alanine to enhance specificity to a target site, and is also referred to as a high-fidelity Cas9 mutant in the present specification. For example, the high specificity Cas9 mutant is a mutant in which at least one amino acid selected from the group consisting of K848, K1003, R1060, N497, R661, Q695, and Q926 is replaced with alanine, based on the amino acid sequence ofStreptococcuspyogenes Cas9 protein (SEQ ID NO: RTI ID = 0.0 > Cas9 < / RTI > In one embodiment, the high specificity Cas9 mutant is Cas9- 1. 1 in which the K848A, K1003A, and R1060A mutations are introduced into theStreptococcuspyogenes Cas9 protein (SEQ ID NO: 4), or Cas9-1.1 in which the N497A, R661A, Q695A, and Q926A mutations are introduced. HF1, or a combination thereof.
본 명세서에서, 가이드 RNA의 매칭된 5' 뉴클레오타이드는 가이드 RNA가 표적하는 표적 서열의 가장 5' 말단에 위치하는 뉴클레오타이드 (5' 뉴클레오타이드라 칭함)와 부합하는 (PAM 서열이 위치하는 가닥의 표적 서열의 5' 뉴클레오타이드와 일치하는) 염기를 포함하는 뉴클레오타이드를 의미한다. 또한 미스매치 5' 뉴클레오타이드는 가이드 RNA가 표적하는 표적 서열의 5' 뉴클레오타이드와 부합하지 않는 (PAM 서열이 위치하는 가닥의 표적 서열의 5' 뉴클레오타이드와 불일치하는) 염기를 포함하는 뉴클레오타이드를 의미한다.In the present specification, the matched 5 'nucleotides of the guide RNA correspond to nucleotides (referred to as 5' nucleotides) located at the 5'end of the target sequence targeted by the guide RNA (the nucleotide sequence of the target sequence of the strand in which the PAM sequence is located Quot; 5 " nucleotides) nucleotides. Also, a mismatch 5 'nucleotide refers to a nucleotide comprising a base that does not match the 5' nucleotide of the target sequence targeted by the guide RNA (inconsistent with the 5 'nucleotide of the target sequence of the strand where the PAM sequence is located).
다른 예는 상기 유전자 교정 방법에 의하여 교정된 유전자를 포함하는 유전자 변형 세포를 제공한다.Another example provides a genetically modified cell comprising the gene corrected by the above gene correction method.
다른 예는 상기 유전자 변형 세포로부터 얻어진 유전자 변형 동물을 제공한다.Another example provides a transgenic animal obtained from said transgenic cell.
상기 방법, 복합체 및 조성물은 진핵 세포 (예컨대, 분리된 인간 세포, 또는 인간을 제외한 진핵 동물 또는 진핵 식물의 세포) 또는 진핵 유기체 (예컨대, 인간, 또는 인간을 제외한 인간을 제외한 진핵 동물 또는 진핵 식물)에 적용되는 것일 수 있다.The methods, complexes, and compositions can be used to treat eukaryotic cells (e. G., Isolated human cells, or eukaryotic or eukaryotic cells other than humans) or eukaryotic organisms (e. G., Human, As shown in FIG.
다른 예는 (1) 가이드 RNA (예컨대, sgRNA) 및 (2) 상기 가이드 RNA의 5' 말단에 융합된 자가-절단 활성을 갖는 RNA 절단효소 또는 1 내지 6개의 tRNA를 포함하는, 융합 RNA 분자를 제공한다.Another example is a fusion RNA molecule comprising (1) a guide RNA (e. G., SgRNA) and (2) an RNA cleavage enzyme having a self-cleaving activity fused to the 5 ' to provide.
다른 예는 가이드 RNA (예컨대, sgRNA)의 5' 말단, 3' 말단, 또는 양 말단에 자가-절단 활성을 갖는 RNA 절단효소 또는 1 내지 6개의 tRNA를 융합시키는 단계를 포함하는, 표적 서열과 매칭된 5' 말단 뉴클레오타이드를 포함하는 가이드 RNA의 제조 방법을 제공한다. 상기 제조 방법에서 융합시키는 단계는 가이드 RNA (예컨대, sgRNA)의 암호화 DNA와 자가-절단 활성을 갖는 RNA 절단효소 또는 1 내지 6개의 tRNA를 암호화하는 DNA를 하나의 벡터에서 발현시키는 단계를 포함할 수 있다.Another example is to match a target sequence comprising a step of fusing an RNA cleaving enzyme or one to six tRNAs having a self-cleaving activity at the 5'-, 3'-, or both ends of a guide RNA (e.g., sgRNA) Lt; / RTI > nucleotides and a 5 ' -terminal nucleotide. The step of fusing in the above production method may include the step of expressing in a vector a DNA coding for an encoding DNA of a guide RNA (for example, sgRNA) and an RNA cleaving enzyme having self-cleavage activity or 1 to 6 tRNA have.
상기 자가-절단 활성을 갖는 RNA 절단효소(리보자임)은 해머해드 리보자임 (hammerhead ribozyme; 예컨대, Type I hammerhead ribozyme, Type II hammerhead ribozyme, TypeIII hammerhead ribozyme 등), VS (Varkud satellite) 리보자임, 리드자임 (Leadzyme), 헤어핀 리보자임 (hairpin ribozyme) 등으로 이루어진 군에서 선택된 1종 이상일 수 있으나, 이에 제한되는 것은 아니다.(Ribozyme) having the self-cleaving activity may be selected from the group consisting of hammerhead ribozyme (for example, type I hammerhead ribozyme, type II hammerhead ribozyme, type III hammerhead ribozyme and the like), VS (Varkud satellite) ribozyme, A hairpin ribozyme, and the like, but the present invention is not limited thereto.
다른 예는 매칭된 5' 말단 뉴클레오타이드를 포함하는 가이드 RNA를 상기 고특이성 Cas9 변이체와 함께 진핵 세포 또는 진핵 유기체에 도입시키는 단계를 포함하는, 상기 고특이성 Cas9 변이체의 유전자 교정 효율 증진 방법을 제공한다. 상기 유전자 교정 효율 증진은 온-타겟 부위에서의 유전자 교정 효율 (예컨대, indel 빈도) 증가 및/또는 오프-타겟 부위에서의 유전자 교정 효율 (예컨대, indel 빈도) 감소를 의미하는 것일 수 있다.Another example provides a method for enhancing the efficiency of gene purification of said high specificity Cas9 variants, comprising introducing a guide RNA comprising matched 5 'terminal nucleotides together with said high specificity Cas9 variant into a eukaryotic or eukaryotic organism. The enhancement of the gene correction efficiency may refer to an increase in the gene correction efficiency (e.g., indel frequency) at the on-target site and / or a decrease in the gene correction efficiency (e.g., indel frequency) at the off-target site.
상기 진핵 세포는 분리된 인간 세포, 또는 인간을 제외한 진핵 동물 세포 또는 진핵 식물의 세포일 수 있고, 상기 진핵 유기체는 인간, 또는 인간을 제외한 인간을 제외한 진핵 동물 또는 진핵 식물일 수 있다.The eukaryotic cell may be an isolated human cell, or a eukaryotic animal cell, or a cell of an eukaryotic plant other than a human, and the eukaryotic organism may be a human, or a eukaryotic animal or eukaryotic plant other than a human, but not a human.
본 명세서에 사용된 바로서, 용어 "유전자 교정 (gene editing)"은 표적 유전자 내의 표적 부위에 이중가닥 절단 (double-stranded DNA cleavage)을 발생시켜서 하나 이상의 뉴클레오타이드의 변이 (결실, 치환, 및/또는 삽입 등)를 유발하는 작용을 의미한다. 일 예에서, 상기와 같은 유전자 교정은 표적 부위에 종료코돈을 생성시키거나, 야생형과 다른 아미노산을 코딩하는 코돈을 생성시킴으로써, 표적 유전자를 불활성화 (knock-out)시키거나, 단백질을 생성하지 않는 비코딩 DNA 서열에 변이를 도입하는 등 다양한 형태일 수 있으나, 이에 제한되는 것은 아니다.As used herein, the term " gene editing " refers to a method of generating double-stranded DNA cleavage at a target site in a target gene, resulting in mutation (deletion, substitution, and / or deletion) of one or more nucleotides Insertion, etc.). In one example, genetic correction as described above can be accomplished by generating a termination codon at the target site, or by generating a codon that codes for a different amino acid than the wild type, thereby knocking out the target gene, But are not limited to, introducing mutations into non-coding DNA sequences.
본 명세서에서, 상기 유전자 교정은 생체 외 (in vitro) 또는 생체 내 (in vivo)에서 수행되는 것일 수 있다.In this specification, the gene correction may be performed in vitro or in vivo.
본 명세서에 사용된 바로서, 용어 "염기 서열"은 해당 염기를 포함하는 뉴클레오타이드의 서열을 의미하는 것으로, 뉴클레오타이드 서열 또는 핵산 서열과 동일한 의미로 사용될 수 있다.As used herein, the term " base sequence " means a sequence of a nucleotide comprising the base, which may be used in the same sense as a nucleotide sequence or a nucleic acid sequence.
본 명세서에 사용된 바로서,As used herein, "
용어 '표적 유전자 (target gene)'는 유전자 교정의 대상이 되는 유전자를 의미하고,The term " target gene " means a gene to be subjected to gene correction,
용어 '표적 부위 (target site or target region)'는 표적 유전자 내의 Cas9에 의한 유전자 교정이 일어나는 부위를 의미하는 것으로, 일 예에서 표적 유전자 내의 Cas9이 인식하는 서열 (PAM 서열)의 5' 말단 및/또는 3' 말단에 인접하여 위치하고, 최대 길이가 약 50bp 또는 약 40bp인 유전자 부위 (이중 가닥 또는 이중 가닥 중 어느 하나의 단일 가닥)를 의미하고,The term " target site or target region " refers to a site where Cas9-induced gene rearrangement occurs in the target gene. In one example, the 5 'end and / Or a gene site (a single strand of either double or double strand) located adjacent to the 3 ' end and having a maximum length of about 50 bp or about 40 bp,
'표적 서열 (target sequence)'는 표적 유전자 또는 표적 유전자의 표적 부위 내의 가이드 RNA가 혼성화하는 약 15 내지 약 30개, 약 15 내지 약 35개, 약 17 내지 약 23개, 또는 약 18개 내지 약 22개, 예컨대, 약 20개의 뉴클레오타이드(nt)를 포함하는 부위의 염기서열일 수 있다.A "target sequence" refers to a sequence of about 15 to about 30, about 15 to about 35, about 17 to about 23, or about 18 to about about 30 hybridizing guide RNAs in a target region of a target gene or target gene 22 nucleotides (nt), for example, about 20 nucleotides (nt).
또한, 가이드 RNA에 포함된 용어 '표적화 서열 (targeting sequence)'은 표적 부위 내의 연속하는 약 15 내지 약 30개, 약 15 내지 약 35개, 약 17 내지 약 23개, 또는 약 18개 내지 약 22개, 예컨대, 약 20개의 뉴클레오타이드(nt)를 포함하는 부위의 염기서열과 상보적인 염기서열을 포함하는(혼성화 가능한) 가이드 RNA의 부위일 수 있다. 상기 표적화 서열과 상보적인 염기서열을 포함하는 표적 부위의 염기서열을 '표적 서열 (target sequence)'이라고 칭할 수 있으며, 상기 표적 서열은 RNA-가이드 뉴클레아제가 인식하는 PAM 서열의 5' 말단 및/또는 3' 말단에 인접하여 위치하는 연속하는 약 15nt 내지 약 30nt, 약 15nt 내지 약 25nt, 약 17nt 내지 약 23nt, 또는 약 18nt 내지 약 22 nt, 예컨대, 약 20nt 길이의 염기서열을 의미할 수 있다.The term "targeting sequence" included in the guide RNA also includes about 15 to about 30 consecutive, about 15 to about 35 consecutive, about 17 to about 23 consecutive, or about 18 to about 22 consecutive, (Hybridizable) guide RNA comprising a nucleotide sequence complementary to a nucleotide sequence of a region containing about 20 nucleotides (nt), for example, a nucleotide sequence of about 20 nucleotides (nt). The nucleotide sequence of the target region including the nucleotide sequence complementary to the targeting sequence may be referred to as a 'target sequence', and the target sequence may be the 5 'end and / or the 5' end of the PAM sequence recognized by the RNA- Or from about 15 nt to about 30 nt, from about 15 nt to about 25 nt, from about 17 nt to about 23 nt, or from about 18 nt to about 22 nt, such as about 20 nt in length, contiguous to the 3 ' .
상기 Cas9 단백질은 표적 유전자의 특정 서열 (PAM)을 인식하고 뉴클레오티드 절단 활성을 가져 표적 유전자에서 인델 (insertion and/or deletion, Indel)을 야기할 수 있는 모든 Cas9들 중에서 선택된 1종 이상일 수 있다.The Cas9 protein recognizes a specific sequence (PAM) of a target gene and has a nucleotide-cleaving activity and can be at least one selected from among all casases capable of inducing indel (insertion and / or deletion, Indel) in a target gene.
상기 Cas9 단백질은 원핵 세포, 및/또는 인간 세포를 비롯한 동식물 세포 (예컨대, 진핵 세포)의 유전체에서 특정 염기서열을 인식해 이중나선절단 (double strand break, DSB)을 일으킬 수 있다. 상기 이중나선절단은 DNA의 이중 나선을 잘라, 둔단 (blunt end) 또는 점착종단 (cohesive end)을 생성시킬 수 있다. DSB는 세포 내에서 상동재조합 (homologous recombination) 또는 비상동재접합 (non-homologous end-joining, NHEJ) 기작에 의해 효율적으로 수선될 수 있는데, 이 과정에 소망하는 변이를 표적 위치에 도입할 수 있다.The Cas9 protein recognizes a specific nucleotide sequence in the genome of animal and plant cells (e. G., Eukaryotic cells) including prokaryotes and / or human cells and can cause double strand breaks (DSB). The double helix cleavage can cut the double helix of DNA to produce a blunt end or a cohesive end. DSBs can be efficiently repaired by homologous recombination or non-homologous end-joining (NHEJ) mechanisms in cells, where desired mutations can be introduced into the target site.
상기 Cas9 단백질은 유전체 DNA의 표적 부위로 안내하기 위한 표적 DNA 특이적 가이드 RNA와 함께 사용된다. 상기 가이드 RNA는 생체 외 (in vitro) 또는 세포 외에서 전사(transcribed; 예컨대 올리고뉴클레오티드 이중가닥 또는 플라스미드 주형으로부터 전사)되거나, 생체 내 또는 세포 내에서 재조합 벡터 (발현 벡터)에 의하여 재조합적으로 생산된 것일 수 있으나, 이에 제한되지 않는다. 상기 Cas9 단백질은, 생체 (또는 세포) 외에서 또는 생체(세포) 내 전달 후, 가이드 RNA와 복합체를 형성하여 리보핵산 단백질 (RNP) 형태로 작용할 수 있다.The Cas9 protein is used in conjunction with a target DNA specific guide RNA to guide the target region of the genomic DNA. The guide RNA may be either in vitro or extracellularly transcribed (e.g., transcribed from an oligonucleotide double strand or plasmid template), recombinantly produced in vivo or in a cell by a recombinant vector (expression vector) But is not limited thereto. The Cas9 protein may function as a ribonucleic acid protein (RNP) by forming a complex with a guide RNA outside the living body (or cell) or in vivo (cellular) delivery.
Cas9 단백질은 CRISPR/Cas 시스템의 주요 단백질 구성 요소로, 활성화된 엔도뉴클레아제 또는 nickase를 형성할 수 있는 단백질이다.Cas9 protein is a major protein component of the CRISPR / Cas system and is capable of forming an activated endonuclease or nickase.
Cas9 단백질 또는 유전자 정보는 NCBI (National Center for Biotechnology Information)의 GenBank와 같은 공지의 데이터 베이스에서 얻을 수 있다. 예컨대, 상기 Cas9 단백질은,Cas9 protein or gene information can be obtained from a known database such as GenBank of National Center for Biotechnology Information (NCBI). For example, the < RTI ID = 0.0 &
스트렙토코커스 sp. (Streptococcus sp.), 예컨대, 스트렙토코커스 피요게네스 (Streptococcuspyogenes) 유래의 Cas9 단백질 (예컨대, SwissProt Accession number Q99ZW2(NP_269215.1));Streptococcus sp. (E.g., SwissProt Accession number Q99ZW2 (NP_269215.1)) fromStreptococcus sp., Such asStreptococcuspyogenes ;
캄필로박터 속, 예컨대, 캄필로박터 제주니 (Campylobacterjejuni) 유래의 Cas9 단백질;Campylobacter genus, for example, Campylobacter Jeju Needle(Campylobacter Cas9 protein derived fromjejuni ;
스트렙토코커스 속, 예컨대, 스트렙토코커스 써모필러스 (Streptococcus thermophiles) 또는 스트렙토코커스 아우레우스 (Streptocuccusaureus) 유래의 Cas9 단백질;Streptococcus genus, for example, Streptococcus Thermo filler's(Streptococcus thermophiles) or Streptococcus aureus(Streptocuccus Cas9 protein fromaureus ;
네이세리아 메닝기디티스 (Neisseria meningitidis) 유래의 Cas9 단백질;Cas9 protein fromNeisseria meningitidis ;
파스테우렐라 (Pasteurella) 속, 예컨대, 파스테우렐라 물토시다 (Pasteurella multocida) 유래의 Cas9 단백질;Paz Chateau Pasteurella(Pasteurella) in, for example, Pas Chateau Pasteurella water Toshio the(Pasteurella multocida) derived Cas9 protein;
프란시셀라 (Francisella) 속, 예컨대, 프란시셀라 노비시다 (Francisella novicida) 유래의 Cas Cas9 단백질Fran when cellar(Francisella) in, for example, when Francisco Cellar Novi Let(Francisellanovicida) derived from Cas protein Cas9
등으로 이루어진 군에서 선택된 하나 이상일 수 있으나, 이에 제한되는 것은 아니다.And the like, but the present invention is not limited thereto.
상기 Cas9 단백질은 미생물에서 분리된 것 또는 재조합적 방법 또는 합성적 방법 등과 같이 인위적 또는 비자연적 생산된 것(non-naturally occurring)일 수 있다. 상기 Cas9 단백질은 in vitro에서 미리 전사된 mRNA 또는 미리 생산된 단백질 형태, 또는 표적 세포 또는 생체 내에서 발현하기 위하여 재조합 벡터에 포함된 형태로 사용될 수 있다. 일 예에서, Cas9 단백질은 재조합 DNA(Recombinant DNA; rDNA)에 의하여 만들어진 재조합 단백질일 수 있다. 재조합 DAN는 다양한 유기체로부터 얻어진 이종 또는 동종 유전 물질을 포함하기 위하여 분자 클로닝과 같은 유전자 재조합 방법에 의하여 인공적으로 만들어진 DNA 분자를 의미한다. 예컨대, 재조합 DNA를 적절한 유기체에서 발현시켜 Cas9 단백질을 생산 (invivo 또는in vitro)하는 경우, 재조합 DNA는 제조하고자 하는 단백질을 코딩 하는 코돈들 중에서 상기 유기체에 발현하기에 최적화된 코돈을 선택하여 재구성된 뉴클레오타이드 서열을 갖는 것일 수 있다.The Cas9 protein may be isolated from microorganisms or artificially or non-naturally occurring such as recombinant or synthetic methods. The Cas9 protein may be used in the form of a mRNA previously transcribed in vitro or a pre-produced protein form, or a form contained in a recombinant vector for expression in a target cell or in vivo. In one example, the Cas9 protein may be a recombinant protein made by recombinant DNA (rDNA). Recombinant DAN refers to a DNA molecule artificially created by genetic recombination methods, such as molecular cloning, to include heterologous or homologous genetic material obtained from a variety of organisms. For example, when the recombinant DNA is expressed in an appropriate organism to produce Cas9 protein (invivo orin vitro ), the recombinant DNA can be obtained by selecting a codon optimized for expression in the organism among the codons encoding the protein to be produced, RTI ID = 0.0 > nucleotide < / RTI >
본 명세서에서 사용된 상기 Cas9 단백질은 변이된 형태의 변이 Cas9일 수 있다. 상기 변이 Cas9 단백질은 DNA 이중 가닥을 절단하는 엔도뉴클레아제 활성을 상실하도록 변이된 것을 의미할 수 있으며, 예컨대, 엔도뉴클레아제 활성을 상실하고 니카아제 활성을 갖도록 변이된 변이 표적특이적 뉴클레아제 및 엔도뉴클레아제 활성과 니카아제 활성을 모두 상실하도록 변이된 변이 표적특이적 뉴클레아제 중에서 선택된 1종 이상일 수 있다. 상기 변이 Cas9 단백질이 니카아제 활성을 갖는 것인 경우, 상기 디아미나제에 의한 염기변환(예컨대, 시티딘이 우라딘으로 변환)과 동시 또는 순서와 무관하게 순차적으로, 상기 염기 변환이 일어난 가닥 또는 그 반대 가닥 (예컨대, 염기 변환이 일어난 가닥의 반대 가닥)에서 nick이 도입될 수 있다 (예컨대, PAM 서열의 5' 말단 방향으로 3번째 뉴클레오타이드와 4번째 뉴클레오타이드 사이에 nick이 도입됨). 이와 같은 Cas9 단백질의 변이 (예컨대, 아미노산 치환 등)는 적어도 뉴클레아제의 촉매 활성 도메인 (예컨대, RuvC 촉매 도메인)에서 일어나는 것일 수 있다. 일 예에서, 상기 Cas9 단백질이 스트렙토코커스 피요젠스 유래 Cas9 단백질 (SwissProt Accession number Q99ZW2(NP_269215.1); 서열번호 4)인 경우, 상기 변이는 촉매 활성을 갖는 아스파르트산 잔기 (catalytic aspartate residue; 예컨대, 서열번호 4의 경우 10번째 위치의 아스파르트산 (D10) 등), 서열번호 4의 762번째 위치의 글루탐산 (E762), 840번째 위치의 히스티딘 (H840), 854번째 위치의 아스파라긴 (N854), 863번째 위치의 아스파라긴 (N863), 986번째 위치의 아스파르트산 (D986) 등으로 이루어진 군에서 선택된 하나 이상 임의의 다른 아미노산으로 치환된 돌연변이를 포함할 수 있다. 이 때, 치환되는 임의의 다른 아미노산은 알라닌 (alanine)일 수 있지만, 이에 제한되지 않는다.As used herein, the Cas9 protein may be a mutated form of Cas9. The mutated Cas9 protein may mean that the mutated Cas9 protein is mutated to lose endonuclease activity to cleave the double strand of DNA. For example, a mutant that has lost endonuclease activity and has been mutated to have a niacase activity, Specific nucleases and variant target specific nucleases that are mutated so as to lose both endo-nuclease activity and niacase activity. When the mutated Cas9 protein has a niacase activity, it is preferable that the nucleotide conversion is carried out either sequentially or sequentially with the base conversion by the diaminase (for example, converting cytidine to uradine) A nick can be introduced at the opposite strand (e.g., the opposite strand of the strand where the base conversion takes place) (e.g. nick introduced between the third and fourth nucleotides in the 5 'terminal direction of the PAM sequence). Such a mutation of the Cas9 protein (e.g., amino acid substitution, etc.) may be that occurring at least in the catalytic domain of the nuclease (e.g., the RuvC catalytic domain). In one example, when the Cas9 protein is a Cas9 protein from Streptococcus pyoensis (SwissProt Accession number Q99ZW2 (NP_269215.1); SEQ ID NO: 4), the mutation may comprise a catalytic aspartate residue Aspartic acid (D10) at
다른 예에서, 상기 변이 Cas9 단백질은 야생형 Cas9 단백질과 상이한 PAM 서열을 인식하도록 변이된 것일 수 있다. 예컨대, 상기 Cas9 단백질은 스트렙토코커스 피요젠스 유래 Cas9 단백질의 1135번째 위치의 아스파르트산 (D1135), 1335번째 위치의 아르기닌 (R1335), 및 1337번째 위치의 트레오닌 (T1337) 중 하나 이상, 예컨대 3개 모두가 다른 아미노산으로 치환되어, 야생형 Cas9의 PAM 서열 (NGG)와 상이한 NGA (N은 A, T, G, 및 C 중에서 선택된 임의의 염기임)을 인식하도록 변이된 것일 수 있다.In another example, the mutated Cas9 protein may be mutated to recognize a PAM sequence that differs from the wild-type Cas9 protein. For example, the Cas9 protein may contain at least one of the aspartic acid (D1135) at position 1135 of arginine (R1335) at position 1335, and threonine (T1337) at position 1337 of Cas9 protein from Streptococcus pyojens May be replaced with other amino acids to recognize an NGA (N is any base selected from A, T, G, and C) that differs from the PAM sequence (NGG) of wild-type Cas9.
일 예에서, 상기 변이 Cas9 단백질은 스트렙토코커스 피요젠스 유래 Cas9 단백질의 아미노산 서열 (서열번호 4) 중,In one example, the mutated Cas9 protein is selected from the amino acid sequence (SEQ ID NO: 4) of the Cas9 protein derived from Streptococcus pyoensens,
(1) D10, H840, 또는 D10 + H840;(1) D10, H840, or D10 + H840;
(2) D1135, R1335, T1337, 또는 D1135 + R1335 + T1337; 또는(2) D1135, R1335, T1337, or D1135 + R1335 + T1337; or
(3) (1)과 (2) 잔기 모두(3) the residues of both (1) and (2)
에서 아미노산 치환이 일어난 것일 수 있다.Lt; RTI ID = 0.0 > amino acid < / RTI >
본 명세서에 사용된 바로서, 상기 '다른 아미노산'은, 알라닌, 이소류신, 류신, 메티오닌, 페닐알라닌, 프롤린, 트립토판, 발린, 아스파라긴산, 시스테인, 글루타민, 글리신, 세린, 트레오닌, 티로신, 아스파르트산, 글루탐산, 아르기닌, 히스티딘, 라이신, 상기 아미노산들의 공지된 모든 변형체 중에서, 야생형 단백질이 원래 변이 위치에 갖는 아미노산을 제외한 아미노산들 중에서 선택된 아미노산을 의미한다. 일 예에서, 상기 '다른 아미노산'은 알라닌, 발린, 글루타민, 또는 아르기닌일 수 있다.As used herein, the 'other amino acids' include, but are not limited to, alanine, isoleucine, leucine, methionine, phenylalanine, proline, tryptophan, valine, aspartic acid, cysteine, glutamine, glycine, serine, threonine, tyrosine, aspartic acid, Arginine, histidine, lysine, and any of the known variants of the above amino acids, amino acids other than the amino acids that the wild-type protein originally has at the mutation position. In one example, the 'other amino acid' may be alanine, valine, glutamine, or arginine.
일 예에서, 상기 변이 Cas9 단백질은 엔도뉴클레아제 활성을 상실(예컨대, 니카아제 활성을 갖거나, 엔도뉴클레아제 활성 및 니카아제 활성을 모두 상실)한 변형 Cas9 단백질, 또는 야생형 Cas9과 상이한 PAM 서열을 인식하는 것일 수 있다. 예컨대, 상기 변이 Cas9 단백질은, 스트렙토코커스 피요제네스 (Streptococcus pyogenes) 유래의 Cas9 단백질(서열번호 4)에 있어서,In one example, the mutated Cas9 protein is a modified Cas9 protein that has lost endonuclease activity (e. G., Has a niacase activity, or has lost both endo-nuclease activity and niacase activity), or a PAM It may be to recognize the sequence. For example, the mutated Cas9 protein is a Cas9 protein (SEQ ID NO: 4) derived fromStreptococcus pyogenes ,
(1) D10 또는 H840 위치에 돌연변이 (예컨대, 다른 아미노산으로의 치환)가 도입되어 엔도뉴클레아제 활성이 상실되고 니카아제 활성을 갖는 변형 Cas9, 또는 스트렙토코커스 피요젠스 (Streptococcuspyogenes) 유래의 Cas9 단백질에 D10 및 H840 위치에 모두 돌연변이 (예컨대, 다른 아미노산으로의 치환)가 도입되어 엔도뉴클레아제 활성 및 니카아제 활성을 모두 상실한 변형 Cas9 단백질;(1) a strain Cas9 having a mutation (for example, substitution with another amino acid) at the position of D10 or H840 to lose endonuclease activity and having a niacase activity, or a Cas9 protein derived fromStreptococcuspyogenes A modified Cas9 protein in which both mutations (e.g., substitution with another amino acid) are introduced at the positions of D10 and H840 to lose both endonuclease activity and niacase activity;
(2) D1135, R1335 및 T1337 중에서 하나 이상 또는 이들 모두에 돌연변이(예컨대, 다른 아미노산으로의 치환)가 도입되어 야생형과 상이한 PAM 서열을 인식하는 변형 Cas9 단백질; 또는(2) a modified Cas9 protein recognizing a PAM sequence different from the wild type by introducing mutations (e.g., substitution with another amino acid) in one or more of D1135, R1335, and T1337; or
(3) (1) 및 (2)의 돌연변이가 모두 도입되어 니카아제 활성을 갖고 야생형과 상이한 PAM 서열을 인식하거나, 엔도뉴클레아제 활성 및 니카아제 활성을 모두 상실하고 야생형과 상이한 PAM 서열을 인식하는 변형 Cas9 단백질(3) Mutations of (1) and (2) are all introduced to recognize PAM sequences having a nicase activity and different from wild type, or to lose endonuclease activity and niacase activity and recognize PAM sequences that are different from wild type Strain Cas9 protein
일 수 있다.Lt; / RTI >
예컨대, 상기 CAs9 단백질의 D10 위치에서의 돌연변이는 D10A 돌연변이 (Cas9 단백질의 아미노산 중 10번째 아미노산인 D가 A로 치환된 돌연변이를 의미함; 이하, Cas9에 도입된 돌연변이는 동일한 방법으로 표기됨)일 수 있고, 상기 H840 위치에서의 돌연변이는 H840A 돌연변이일 수 있으며, D1135, R1335, 및 T1337 위치에서의 돌연변이는 각각 D1135V, R1335Q, 및T1337R일 수 있다.For example, the mutation at the D10 position of the CAs9 protein is a D10A mutation (a mutation in which the 10th amino acid of the amino acid of the Cas9 protein is substituted with A; hereinafter, the mutation introduced into Cas9 is represented by the same method) And the mutation at the H840 position may be the H840A mutation and the mutation at the D1135, R1335, and T1337 positions may be D1135V, R1335Q, and T1337R, respectively.
상기 Cas9 단백질은 단백질 형태, 이를 코딩하는 핵산 분자 (예컨대, DNA 또는 mRNA), 가이드 RNA와 결합된 리보핵산 단백질, 상기 리보핵산 단백질을 암호화하는 핵산 분자, 또는 상기 핵산 분자를 포함하는 재조합 벡터의 형태로 사용될 수 있다.The Cas9 protein may be in the form of a protein, a nucleic acid molecule (for example, DNA or mRNA) encoding the same, a ribonucleic acid protein bound to a guide RNA, a nucleic acid molecule encoding the ribonucleic acid protein, or a form of a recombinant vector .
상기 Cas9 단백질을 암호화하는 핵산 분자는 핵 내로 전달, 작용, 및/또는 핵 내에서 발현될 수 있는 형태일 수 있다.The nucleic acid molecule encoding the Cas9 protein may be in a form that can be delivered into the nucleus, acted on, and / or expressed in the nucleus.
상기 Cas9 단백질은 세포 내로 도입되기에 용이한 형태일 수 있다. 일 예로, 상기 Cas9 단백질 또는 이를 암호화하는 유전자는 세포 침투 펩타이드 및/또는 단백질 전달 도메인 (protein transduction domain) 또는 이를 암호화하는 유전자와 연결될 수 있다. 상기 단백질 전달 도메인은 폴리-아르기닌 또는 HIV 유래의 TAT 단백질일 수 있으나, 이에 제한되지 않는다. 세포 침투 펩타이드 또는 단백질 전달 도메인은 상기 기술된 예 외에도 다양한 종류가 당업계에 공지되어 있으므로, 당업자는 상기 예에 제한되지 않고 다양한 예를 적용할 수 있다.The Cas9 protein may be in a form that is easy to be introduced into cells. For example, the Cas9 protein or the gene encoding the Cas9 protein may be linked to a cell penetrating peptide and / or a protein transduction domain or a gene encoding the same. The protein transfer domain may be a poly-arginine or a TAT protein derived from HIV, but is not limited thereto. Various types of cell penetrating peptide or protein transfer domains other than the above-described examples are well known in the art, so that a person skilled in the art can apply various examples without limitation to the above examples.
또한, 상기 Cas9 단백질, 및/또는 이들을 코딩하는 핵산 분자는 핵 위치 신호 (nuclear localization signal, NLS; 예컨대, cccaagaaga agaggaaagtc (서열번호 6))를 추가로 포함할 수 있다. 따라서, 상기 Cas9 단백질 암호화 핵산 분자를 포함하는 발현 카세트는 상기 Cas9 단백질을 발현시키기 위한 프로모터 서열 등의 조절 서열, 및, 임의로, NLS 서열을 추가로 포함할 수 있다. 상기 NLS 서열은 당업계에 잘 알려져 있다.In addition, the Cas9 protein and / or the nucleic acid molecule encoding the same may further comprise a nuclear localization signal (NLS; e.g., cccaagaaga agagga aagtc (SEQ ID NO: 6)). Accordingly, the expression cassette comprising the Cas9 protein-encoding nucleic acid molecule may further comprise a regulatory sequence such as a promoter sequence for expressing the Cas9 protein, and optionally, an NLS sequence. Such NLS sequences are well known in the art.
상기 Cas9 단백질, 및/또는 이를 코딩하는 핵산 분자는 분리 및/또는 정제를 위한 태그 또는 상기 태그를 코딩하는 핵산 서열과 연결될 수 있다. 일 예로, 상기 태그는 His 태그, Flag 태그, S 태그 등과 같은 작은 펩타이드 태그, GST (Glutathione S-transferase) 태그, MBP (Maltose binding protein) 태그 등으로 이루어진 군에서 적절하게 선택될 수 있으나, 이에 제한되지 않는다.The Cas9 protein and / or the nucleic acid molecule encoding the Cas9 protein may be associated with a tag for separation and / or purification, or a nucleic acid sequence encoding the tag. For example, the tag may be appropriately selected from the group consisting of a small peptide tag such as a His tag, a Flag tag, an S tag, etc., a GST (Glutathione S-transferase) tag, an MBP (Maltose binding protein) tag, It does not.
본 명세서에서, 용어 "가이드 RNA (guide RNA)"는 표적 유전자 내의 표적 부위 내의 특이적인 염기 서열 (표적서열)에 혼성화 가능한 표적화 서열을 포함하는 RNA를 의미하며, 생체 외 (in vitro) 또는 생체 (또는 세포) 내에서 Cas9 단백질과 결합하여 이를 표적 유전자 (또는 표적 부위)로 인도하는 역할을 한다.As used herein, the term " guide RNA " refers to an RNA comprising a targeting sequence capable of hybridizing to a specific base sequence (target sequence) in a target site in a target gene, and may be in vitro or in vivo Or cells) and directs them to the target gene (or target site).
상기 가이드 RNA는 복합체를 형성할 Cas9 단백질의 종류 및/또는 그 유래 미생물에 따라서 적절히 선택될 수 있다.The guide RNA may be appropriately selected according to the kind of Cas9 protein to be complexed and / or the microorganism from which it is derived.
예컨대, 상기 가이드 RNA는,For example,
표적 서열과 혼성화 가능한 부위 (표적화 서열)을 포함하는 CRISPR RNA (crRNA);CRISPR RNA (crRNA) comprising a site capable of hybridizing with a target sequence (targeting sequence);
Cas9 단백질과 상호작용하는 부위를 포함하는 trans-activating crRNA (tracrRNA); 및Trans- activating crRNA (tracrRNA) comprising a site that interacts with the Cas9 protein; And
상기 crRNA 및 tracrRNA의 주요 부위 (예컨대, 표적화 서열을 포함하는 crRNA 부위 및 뉴클레아제와 상호작용하는 tracrRNA의 부위)가 융합된 형태의 단일 가이드 RNA (single guide RNA; sgRNA)A single guide RNA (sgRNA) in the form of fusion of the major parts of the crRNA and the tracrRNA (for example, a crRNA site including a targeting sequence and a site of a tracrRNA interacting with a nuclease)
로 이루어진 군에서 선택된 1종 이상일 수 있으며,And may be at least one selected from the group consisting of
구체적으로 CRISPR RNA (crRNA) 및trans-activating crRNA (tracrRNA)를 포함하는 이중 RNA (dual RNA), 또는 crRNA 및 tracrRNA의 주요 부위를 포함하는 단일 가이드 RNA (sgRNA)일 수 있다.Specifically a dual RNA comprising CRISPR RNA (crRNA) and atrans- activating crRNA (tracrRNA), or a single guide RNA (sgRNA) comprising a major region of a crRNA and tracrRNA.
상기 sgRNA는 표적 유전자 (표적 부위) 내의 표적 서열과 상보적인 서열 (표적화 서열)을 가지는 부분 (이를 Spacer region, Target DNA recognition sequence, base pairing region 등으로도 명명함) 및 Cas 단백질 결합을 위한 hairpin 구조를 포함할 수 있다. 보다 구체적으로, 표적 유전자 내의 표적서열과 상보적인 서열(표적화 서열)을 포함하는 부분, Cas 단백질 결합을 위한 hairpin 구조, 및 Terminator 서열을 포함할 수 있다. 상기 기술된 구조는 5'에서 3' 순으로 순차적으로 존재하는 것일 수 있으나, 이에 제한되는 것은 아니다. 상기 가이드 RNA가 crRNA 및 tracrRNA의 주요 부분 및 표적 DNA의 상보적인 부분을 포함하는 경우라면 어떠한 형태의 가이드 RNA도 본 발명에서 사용될 수 있다.The sgRNA is a part having a sequence (a targeting sequence) complementary to a target sequence in a target gene (also referred to as a target DNA recognition sequence, a base pairing region, etc.) and a hairpin structure . ≪ / RTI > More specifically, it may include a portion including a sequence complementary to the target sequence in the target gene (targeting sequence), a hairpin structure for cas protein binding, and a terminator sequence. The structures described above may be sequentially present in the order of 5 'to 3', but are not limited thereto. Any type of guide RNA may be used in the present invention if the guide RNA comprises a major portion of the crRNA and tracrRNA and a complementary portion of the target DNA.
예컨대, Cas9 단백질은 표적 유전자 교정을 위하여 두 개의 가이드 RNA, 즉, 표적 유전자의 표적 부위와 혼성화 가능한 뉴클레오타이드 서열을 갖는 CRISPR RNA (crRNA)와 Cas9 단백질와 상호작용하는trans-activating crRNA (tracrRNA; Cas9 단백질과 상호작용함)를 필요로 하며, 이들 crRNA와 tracrRNA는 서로 결합된 이중 가닥 crRNA:tracrRNA 복합체 형태, 또는 링커를 통하여 연결되어 단일 가이드 RNA (single guide RNA; sgRNA) 형태로 사용될 수 있다. 일 예에서,Streptococcus pyogenes 유래의 Cas9 단백질을 사용하는 경우, sgRNA는 적어도 상기 crRNA의 혼성화 가능한 뉴클레오타이드 서열을 포함하는 crRNA 일부 또는 전부와 상기 Cas9의 tracrRNA의 Cas9 단백질와 상호작용하는 부위를 적어도 포함하는 tracrRNA 일부 또는 전부가 뉴클레오타이드 링커를 통하여 헤어핀 구조 (stem-loop 구조)를 형성하는 것일 수 있다 (이 때 뉴클레오타이드 링커가 루프 구조에 해당할 수 있음).For example, the Cas9 protein contains two guide RNAs, namely, a CRISPR RNA (a crRNA) having a nucleotide sequence capable of hybridizing with a target region of a target gene and atrans- activating crRNA (a casp These crRNAs and tracrRNAs can be used in the form of a double-stranded crRNA: tracrRNA complex linked to each other or in the form of a single guide RNA (sgRNA) linked through a linker. In one example, when a Cas9 protein derived fromStreptococcus pyogenes is used, the sgRNA comprises at least a portion of the crRNA comprising the hybridizable nucleotide sequence of the crRNA, and a portion of the tracrRNA portion comprising at least a site that interacts with the Cas9 protein of the cas9 tracrRNA Or all may form a hair-pin structure (stem-loop structure) through the nucleotide linker (the nucleotide linker may correspond to a loop structure).
상기 가이드 RNA, 구체적으로 crRNA 또는 sgRNA는 표적 유전자 내 표적 서열과 상보적인 서열(표적화 서열)을 포함하며, crRNA 또는 sgRNA의 업스트림 부위, 구체적으로 sgRNA 또는 dualRNA의 crRNA의 5' 말단에 하나 이상, 예컨대, 1-10개, 1-5개, 또는 1-3개의 추가의 뉴클레오티드를 포함할 수 있다. 상기 추가의 뉴클레오티드는 구아닌 (guanine, G)일 수 있으나, 이에 제한되는 것은 아니다.The guide RNA, specifically a crRNA or an sgRNA, comprises a sequence complementary to a target sequence in a target gene (targeting sequence), and includes at least one upstream region of a crRNA or sgRNA, specifically at the 5 'end of a sRNA or dualRNA crRNA, , 1-10, 1-5, or 1-3 additional nucleotides. The additional nucleotide may be, but is not limited to, guanine (G).
상기 가이드 RNA의 구체적 서열은 Cas9의 종류 (즉, 유래 미생물)에 따라서 적절히 선택할 수 있으며, 이는 이 발명이 속하는 기술 분야의 통상의 지식을 가진 자가 용이하게 알 수 있는 사항이다.The specific sequence of the guide RNA can be appropriately selected according to the kind of Cas9 (i.e., derived microorganism), and it is easily understood by those skilled in the art.
일 예에서, 표적특이적 뉴클레아제로서Streptococcuspyogenes 유래의 Cas9 단백질을 사용하는 경우, crRNA는 다음의 일반식 1로 표현될 수 있다:In one example, when the Cas9 protein fromStreptococcuspyogenes is used as the target specific nuclease, the crRNA may be represented by the following general formula 1:
5'-(Ncas9)l-(GUUUUAGAGCUA)-(Xcas9)m-3' (일반식 1)5 '- (Ncas9 )1 - (GUUUUAGAGCUA) - (Xcas9 )m -3'
상기 일반식 1에서,In the general formula 1,
Ncas9는 표적화 서열, 즉 표적 유전자(target gene)의 표적 부위(target site)의 서열에 따라서 결정되는 부위 (표적 부위의 표적 서열과 혼성화 가능)이며, l은 상기 표적화 서열에 포함된 뉴클레오타이드 수를 나타내는 것으로 15 내지 30, 17 내지 23, 또는 18 내지 22의 정수, 예컨대 20일 수 있고,Ncas9 is a targeting sequence, that is, a site (hybridizable with the target sequence of the target site) determined according to the sequence of the target site of the target gene, l is the number of nucleotides contained in the targeting sequence May be an integer of 15 to 30, 17 to 23, or 18 to 22, for example 20,
상기 표적화 서열의 3' 방향으로 인접하여 위치하는 연속하는 12개의 뉴클레오타이드(GUUUUAGAGCUA) (서열번호 1)를 포함하는 부위는 crRNA의 필수적 부분이고,The site containing the consecutive 12 nucleotides (GUUUUAGAGCUA) (SEQ ID NO: 1) located in the 3 'direction of the targeting sequence is an essential part of the crRNA,
Xcas9는 crRNA의 3' 말단쪽에 위치하는 (즉, 상기 crRNA의 필수적 부분의 3' 방향으로 인접하여 위치하는) m개의 뉴클레오타이드를 포함하는 부위로, m은 8 내지 12의 정수, 예컨대 11일 수 있으며, 상기 m개의 뉴클레오타이드들은 서로 같거나 다를 수 있으며, 각각 독립적으로 A, U, C 및 G로 이루어진 군에서 선택될 수 있다.Xcas9 is a site containing m nucleotides located at the 3 'terminal side of the crRNA (i.e., located adjacent to the 3' direction of the essential part of the crRNA), and m is an integer of 8 to 12, And the m nucleotides may be the same or different from each other, and may be independently selected from the group consisting of A, U, C and G.
일 예에서, 상기 Xcas9는 UGCUGUUUUG (서열번호 2)를 포함할 수 있으나 이에 제한되지 않는다.In one example, Xcas9 may include, but is not limited to, UGCUGUUUUG (SEQ ID NO: 2).
또한, 상기 tracrRNA는 다음의 일반식 2로 표현될 수 있다:In addition, the tracrRNA can be represented by the following general formula 2:
5'-(Ycas9)p-(UAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC)-3' (일반식 2)5 '- (Ycas9 )p - (UAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC) -3' (Formula 2)
상기 일반식 2에서,In the general formula 2,
60개의 뉴클레오타이드 (UAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC) (서열번호 3)로 표시된 부위는 tracrRNA의 필수적 부분이고,The site represented by 60 nucleotides (UAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC) (SEQ ID NO: 3) is an essential part of the tracrRNA,
Ycas9는 상기 tracrRNA의 필수적 부분의 5' 말단에 인접하여 위치하는 p개의 뉴클레오타이드를 포함하는 부위로, p는 6 내지 20의 정수, 예컨대 8 내지 19의 정수일 수 있으며, 상기 p개의 뉴클레오타이드들은 서로 같거나 다를 수 있고, A, U, C 및 G로 이루어진 군에서 각각 독립적으로 선택될 수 있다.Ycas9 is a site containing p nucleotides located adjacent to the 5 'end of an essential part of the tracrRNA, p may be an integer of 6 to 20, such as an integer of 8 to 19, and the p nucleotides may be the same And may be independently selected from the group consisting of A, U, C and G,
또한, sgRNA는 상기 crRNA의 표적화 서열과 필수적 부위를 포함하는 crRNA 부분과 상기 tracrRNA의 필수적 부분 (60개 뉴클레오타이드)를 포함하는 tracrRNA 부분이 올리고뉴클레오타이드 링커를 통하여 헤어핀 구조 (stem-loop 구조)를 형성하는 것일 수 있다 (이 때, 올리고뉴클레오타이드 링커가 루프 구조에 해당함). 보다 구체적으로, 상기 sgRNA는 crRNA의 표적화 서열과 필수적 부분을 포함하는 crRNA 부분과 tracrRNA의 필수적 부분을 포함하는 tracrRNA 부분이 서로 결합된 이중 가닥 RNA 분자에서, crRNA 부위의 3' 말단과 tracrRNA 부위의 5' 말단이 올리고뉴클레오타이드 링커를 통하여 연결된 헤어핀 구조를 갖는 것일 수 있다.In addition, the sgRNA includes a crRNA portion including the targeting sequence of the crRNA and the essential region, and a tracrRNA portion including an essential portion (60 nucleotides) of the tracrRNA form a hair-pin structure (stem-loop structure) through an oligonucleotide linker (At this time, the oligonucleotide linker corresponds to the loop structure). More specifically, the sgRNA is a double-stranded RNA molecule in which a tracrRNA portion including an essential portion of a trcRNA and a crRNA portion including an essential portion of a crRNA and an essential portion of a tracrRNA are bound to each other, Terminus may be a hairpin structure linked through an oligonucleotide linker.
일 예에서, sgRNA는 다음의 일반식 3으로 표현될 수 있다:In one example, the sgRNA can be represented by the following general formula 3:
5'-(Ncas9)l-(GUUUUAGAGCUA)-(올리고뉴클레오타이드 링커)-(UAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC)-3' (일반식 3)5 '- (Ncas9 )1 - (GUUUUAGAGCUA) - (oligonucleotide linker) - (UAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC) -3'
상기 일반식 3에서, (Ncas9)l는 표적화 서열로서 앞서 일반식 1에서 설명한 바와 같다.In the above general formula (3), (Ncas9 )l is a targeting sequence as described in general formula (1).
상기 sgRNA에 포함되는 올리고뉴클레오타이드 링커는 3 내지 5개, 예컨대 4개의 뉴클레오타이드를 포함하는 것일 수 있으며, 상기 뉴클레오타이드들은 서로 같거나 다를 수 있고, A, U, C 및 G로 이루어진 군에서 각각 독립적으로 선택될 수 있다. 일 구체예에서, 상기 올리로뉴클레오타이드 링커는 GAAA의 핵산 서열을 포함하는 것일 수 있으나, 이에 제한되는 것은 아니다.The oligonucleotide linker contained in the sgRNA may include 3 to 5 nucleotides, for example, 4 nucleotides. The nucleotides may be the same or different from each other and are each independently selected from the group consisting of A, U, C and G . In one embodiment, the oligonucleotide linker may comprise, but is not limited to, a nucleic acid sequence of GAAA.
상기 crRNA 또는 sgRNA는 5' 말단 (즉, crRNA의 타겟팅 서열 부위의 5' 말단)에 1 내지 3개의 구아닌(G)을 추가로 포함할 수 있다.The crRNA or sgRNA may further comprise 1 to 3 guanines (G) at the 5 'terminus (i.e., at the 5' terminus of the targeting sequence region of the crRNA).
상기 tracrRNA 또는 sgRNA는 tracrRNA의 필수적 부분(60nt)의 3' 말단에 3개 내지 7개, 3개 내지 5개, 또는 5개 내지 7개의 우라실 (U)을 포함하는 종결부위를 추가로 포함할 수 있다.The tracrRNA or sgRNA may further comprise a terminator region comprising 3 to 7, 3 to 5, or 5 to 7 uracil (U) at the 3 'end of an essential part (60 nt) of the tracrRNA have.
상기 가이드 RNA의 표적 서열은 표적 DNA 상의 PAM (Protospacer Adjacent Motif 서열(S.pyogenes Cas9의 경우, 5'-NGG-3' (N은 A, T, G, 또는 C임))의 5'에 인접하여 위치하는 약 17개 내지 약 23개 또는 약 18개 내지 약22개, 예컨대 20개의 연속하는 핵산 서열일 수 있다.The target sequence of the guide RNA is adjacent to 5 'of the PAM (Protospacer Adjacent Motif sequence on the target DNA (5'-NGG-3' in the case ofS.pyogenes Cas9 (N is A, T, G, or C) For example, from about 17 to about 23 or from about 18 to about 22, such as 20 contiguous nucleic acid sequences.
상기 가이드 RNA의 표적 서열과 혼성화 가능한 가이드 RNA의 표적화 서열은 상기 표적 서열이 위치하는 DNA 가닥 (즉, PAM 서열(5'-NGG-3' (N은 A, T, G, 또는 C임)이 위치하는 DNA 가닥) 또는 이의 상보적인 가닥의 뉴클레오타이드 서열과 50% 이상, 60% 이상, 70% 이상, 80% 이상, 90% 이상, 95% 이상, 99% 이상, 또는 100%의 서열 상보성을 갖는 뉴클레오타이드 서열을 의미하는 것으로, 상기 상보적 가닥의 뉴클레오타이드 서열과 상보적 결합이 가능하다.The targeting sequence of the guide RNA capable of hybridizing with the target sequence of the guide RNA is a DNA strand (i.e., a PAM sequence (5'-NGG-3 '(N is A, T, G, or C) Having at least 50%, at least 60%, at least 70%, at least 80%, at least 90%, at least 95%, at least 99%, or at most 100% complementarity to the nucleotide sequence of the complementary strand Refers to a nucleotide sequence, which is complementary to a nucleotide sequence of the complementary strand.
본 명세서에서, 표적 부위의 핵산 서열(표적 서열)은 표적 유전자의 해당 유전자 부위의 두 개의 DNA 가닥 중 PAM 서열이 위치하는 가닥의 핵산 서열로 표시된다. 이 때, 실제로 가이드 RNA가 결합하는 DNA 가닥은 PAM 서열이 위치하는 가닥의 상보적 가닥일 수 있으므로, 상기 가이드 RNA에 포함된 표적화 서열은, RNA 특성상 T를 U로 변경하는 것을 제외하고, 표적 서열과 동일한 핵산 서열을 가질 수 있다. 따라서, 본 명세서에서, 가이드 RNA의 표적화 서열과 표적 서열은 T와 U가 상호 변경되는 것을 제외하고 동일한 핵산 서열로 표시된다.In the present specification, the nucleic acid sequence (target sequence) of the target site is represented by the nucleic acid sequence of the strand where the PAM sequence is located in the two DNA strands of the corresponding gene site of the target gene. In this case, since the DNA strand to which the guide RNA is actually bound may be a complementary strand of the strand in which the PAM sequence is located, the targeting sequence contained in the guide RNA may include a target sequence And < / RTI > Thus, in the present specification, the targeting sequence and target sequence of the guide RNA are represented by the same nucleic acid sequence except that T and U are mutually modified.
상기 가이드 RNA는 RNA 형태로 사용 (또는 상기 조성물에 포함)되거나, 이를 코딩하는 DNA를 포함하는 플라스미드 형태로 사용 (또는 상기 조성물에 포함)될 수 있다.The guide RNA may be used (or included in the composition) in the form of a plasmid (or included in the composition) in the form of a plasmid containing the DNA encoding the RNA.
본 명세서에서 제공되는 고특이성 Cas9 변이체들의 오프-타겟 부위에 대한 낮은 유전자 교정 활성을 유지하면서 온-타겟 특이적 유전자 교정 활성을 보다 증진시킬 수 있는 기술은 유전공학 분야 및 의약 분야에 광범위하게 적용될 수 있다.A technique capable of further enhancing the on-target specific gene correcting activity while maintaining a low genetic corrective activity against off-target sites of the high specificity Cas9 variants provided herein can be broadly applied in the field of genetic engineering and medicine have.
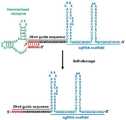
도 1은 고충실도 (high-fidelity) Cas9 변이체들이 non-G 5' nucleotide를 갖는 표적 부위에서의 유전자 교정 활성을 보여주는 것으로,
1a는 야생형 Cas9(Cas9-WT), 조작된 Cas9 변이체들, 및 sgRNA를 모식적으로 보여주며, Cas9-WT와 비교하여, eCas9-1.1 또는 Cas9-HF1에 도입된 알라닌 치환을 각각 청색 또는 적색 별표로 표시하였으며, 적색 삼각형은 절단 위치를 나타내고, GX19 sgRNA는 protospacer (청색줄)와 매칭된 G로 시작하는 것을 나타내고, gX19 sgRNA는 5'-말단에 미스매칭된 G를 갖는 것이고, gX20 sgRNA는 5'-말단에 추가의 구아닌을 포함하는 것이며, PAM (protospacer-adjacent motif)은 적색 줄로 표지된 NGG이며 (H, G이 아님 (A 또는 C 또는 T); D, C가 이님 (A 또는 G 또는 T)),
1b는 HeLa 세포에서 gX19 sgRNA 또는 gX20 sgRNA를 사용하여 얻어진 5' 뉴클레오타이드가 G가 아닌 온-타겟 부위에서의 Indel 빈도를 보여주는 그래프이다 (targeted deep sequencing에 의하여 측정됨, PAM 서열은 파란색으로 표시됨, Error bars, s.e.m.;n = 3).
도 2는 Hammerhead 리보자임-결합 sgRNA를 보여주는 것으로,
2a는 자가-프로세싱 리보자임(self-processing ribozyme)이 융합된 sgRNA를 모식적으로 보여주며, pre-sgRNA는 5' 말단에 Hammerhead (HH) 리보자임을 포함하고, pre-sgRNA는 자가 절단 (self-cleavage)을 거쳐 성숙 sgRNA로 방출되며, 적색 화살표는 자가 절단 위치를 나타내며,
2b는 매칭된 5' 뉴클레오타이드를 갖는 HH 리보자임-융합된 sgRNA (HH-X20) 또는 미스매치 구아노신을 갖는 HH 리보자임-융합된 sgRNA (HH-gX19)을 Cas9-WT 또는 high-fidelity Cas9 변이체와 각각 조합하여 HeLa 에서의 5개의 표적 부위에 대한 유전자 교정 효율을 시험하고, targeted deep sequencing를 사용하여 Indel 빈도를 측정하였으며 (PAM은 청색으로 표시되어 있고, Error bars, s.e.m.;n = 3, Statistical significances were calculated by t-test. *P < 0.05, **P < 0.01),
2c는 5개의 표적 부위에서의 평균 indel 빈도 ± s.e.m.를 보여주는 그래프이며 (*P < 0.05, **P < 0.01),
2d는 HH-X20 sgRNA를 Cas9-WT 또는 Cas9 변이체와 함께 HeLa 세포 내로 공동 형질감염시키고, targeted deep sequencing으로 측정한 온-타겟 및 오프-타겟 부위에서의 Indel 빈도를 나타낸 그래프이다 (PAM 서열은 청색으로 나타내고, 미스매치 염기는 적색으로 표시함; specificity ratio는 Cas9 변이체 및 Cas9-WT를 사용하여 얻어진 오프-타겟 부위에서의 indel 빈도에 대한 온-타겟 부위에서의 indel 빈도의 비율 차이를 배수로 측정한 값 ([Cas9 변이체를 사용하여 얻어진 오프-타겟 부위에서의 indel 빈도에 대한 온-타겟 부위에서의 indel 빈도의 비율]/[Cas9-WT를 사용하여 얻어진 오프-타겟 부위에서의 indel 빈도에 대한 온-타겟 부위에서의 indel 빈도의 비율]); Error bars, s.e.m.;n = 3; mock transfected sample과 비교하여 유의미한 수준의 Indel 빈도는 별표로 표시함 (*P < 0.05, **P < 0.01)).Figure 1 shows the gene correcting activity of high-fidelity Cas9 mutants at the target site with non-G 5 'nucleotide,
1a schematically shows the wild-type Cas9 (Cas9-WT), engineered Cas9 variants, and sgRNA and shows that the alanine substitutions introduced in eCas9-1.1 or Cas9-HF1, respectively, as compared to Cas9-WT, , The red triangle represents the cleavage site, the GX19 sgRNA starts with the G that matches the protospacer (blue line), the gX19 sgRNA has the G-mismatched G at the 5'-end, and the gX20 The sgRNA contains an additional guanine at the 5'-end and a protospacer-adjacent motif (PAM) is a red-labeled NGG (H, G (A or C or T) A or G or T)),
1b is a graph showing the Indel frequency at the on-target site, where the 5 'nucleotide obtained using gX19 sgRNA or gX20 sgRNA in HeLa cells is not G (measured by targeted deep sequencing, the PAM sequence is shown in blue , Error bars, sem;n = 3).
Figure 2 shows the Hammerhead ribozyme-binding sgRNA,
2a schematically shows sgRNA fused with self-processing ribozyme, pre-sgRNA contains Hammerhead (HH) ribozyme at the 5 'end, pre-sgRNA is self- -cleavage) to mature sgRNA, the red arrows indicate self-cleavage sites,
2b is HH ribozyme having a 5 'nucleotide match-fused sgRNA (HH-X20) or has put mismatch guano HH ribozyme-fusedsgRNA (HH-gX 19) Cas9 -WT or high-fidelity Cas9 (PAM is indicated in blue and Error bars, sem;n = 3, PAM is indicated in blue), and the targeted gene sequencing efficiency was measured by targeted deep sequencing. Statistical significances were calculated by t-test. *P <0.05, **P <0.01),
2c is a graph showing the mean indel frequency ± sem at five target sites (*P <0.05, **P <0.01)
2d are graphs showing co-transfection of HH-X20 sgRNA with Cas9-WT or Cas9 variants into HeLa cells and Indel frequency at on-target and off-target sites as measured by targeted deep sequencing (PAM sequence The specificity ratio was calculated as a multiple of the ratio of the indel frequency at the on-target site to the indel frequency at the off-target site obtained using the Cas9 variant and Cas9-WT, The ratio of the indel frequency at the on-target site to the indel frequency at the off-target site obtained using the Cas9 variant / [the indel frequency at the off-target site obtained using Cas9-WT] on-rate of indel incidence in the target region]); as represented in mock transfected significant level of Indel frequency asterisk in comparison withsample (* P <0.05, ** P <0.01); Error bars, sem; n = 3 ).
이하에서는 실시예를 들어 본 발명을 더욱 구체적으로 설명하고자 하나, 이는 예시적인 것에 불과할 뿐 본 발명의 범위를 제한하고자 함이 아니다. 아래 기재된 실시예들은 발명의 본질적인 요지를 벗어나지 않는 범위에서 변형될 수 있음은 당 업자들에게 있어 자명하다.Hereinafter, the present invention will be described in more detail with reference to the following examples, which should not be construed as limiting the scope of the present invention. It will be apparent to those skilled in the art that the embodiments described below may be modified without departing from the essential spirit of the invention.
실시예Example 1: high-fidelity 1: high-fidelityCas9Cas9변이체Mutant 암호화 플라스미드 및 Encoding plasmids andHHHH--리보자임Ribozyme-융합된 sgRNA 암호화 플라스미드의 구축- Construction of fused sgRNA-encoding plasmids
Cas9 변이체를 암호화하는 플라스미드로서,Streptococcuspyogenes Cas9 단백질(서열번호 4)에 K848A, K1003A, 및 R1060A 변이가 도입된 eSpCas9-1.1를 암호화하는 플라스미드 (p3seCas9-1.1; Addgene #104172) 및 N497A, R661A, Q695A, 및 Q926A 변이가 도입된 p3s-Cas9-HF1를 암호화하는 플라스미드 (p3s-Cas9-HF1, Addgene #104173)을 각각 사용하였다.(P3seCas9-1.1; Addgene # 104172) coding for eSpCas9-1.1 with K848A, K1003A and R1060A mutations introduced intoStreptococcuspyogenes Cas9 protein (SEQ ID NO: 4) and N497A, R661A, Q695A , And a plasmid (p3s-Cas9-HF1, Addgene # 104173) encoding p3s-Cas9-HF1 into which the Q926A mutation was introduced were respectively used.
HH(Hammerhead)-리보자임 sgRNA 구조체 (도 2a 참조)는 HH-리보자임 핵산 서열 및 protospacer 서열을 포함하는 어닐링된 올리고뉴클레오타이드를 플라스미드 (pRG2, Addgene #104174; sgRNA가 U6 프로모터 조절 하에서 발현) 내로 ligation함으로써 클로닝하였다.The HH (Hammerhead) -ribozyme sgRNA construct (see Fig. 2a) was prepared by ligation of an annealed oligonucleotide comprising the HH-ribozyme nucleic acid sequence and a protospacer sequence into a plasmid (pRG2, Addgene # 104174; sgRNA expressed under U6 promoter control) .
상기 sgRNA는 다음의 서열을 갖는다:The sgRNA has the following sequence:
5'-(표적 서열)-(GUUUUAGAGCUA; 서열번호 1)-(뉴클레오타이드 링커)-(UAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC; 서열번호 3)-UUUU-3'5 '- (Target Sequence) - (GUUUUAGAGCUA; SEQ ID NO: 1) - (Nucleotide Linker) - (UAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC; SEQ ID NO: 3) -UUUU-3'
(상기 표적 서열은 하기의 표 1에 표시된 표적 부위 서열(20nt)에서 "T"를 "U"로 변환한 서열이며,(The target sequence is a sequence obtained by converting " T " to " U " in the target site sequence (20 nt) shown in Table 1 below,
gX19 sgRNA는 표 1의 모든 표적 부위 서열에 있어서 5' 말단 염기 (밑줄로 표시) 위치에 'G'를 포함하도록 제작된 sgRNA이고,and gX19 sgRNA is the sgRNA produced to include the G '' at the terminal bases (underlined) position, five in all target site sequence shown in Table 1,
X20 sgRNA는 표 1의 각각의 표적 부위 서열의 5' 말단 염기(밑줄로 표시)에 매칭되는 염기를 포함하도록 제작된 sgRNA이며,X20 sgRNA is an sgRNA prepared to include a base that matches the 5 'terminal base (indicated by an underlined letter) of each target site sequence in Table 1,
상기 뉴클레오타이드 링커는 GAAA의 뉴클레오타이드 서열을 가짐).Wherein the nucleotide linker has a nucleotide sequence of GAAA.
실시예Example 2: 세포 배양 및 형질감염 ( 2: Cell culture and transfection (transfectiontransfection))
HeLa cells (ATCC, CCL-2)를 100 units/mL 페니실린, 100 ug(microgram)/mL 스트렙토마이신, 0.1 mM 비필수 아미노산, 및 10%(w/v) 우태아혈? (fetal bovine serum; FBS)이 보충된 Dulbecco's modified Eagle's medium (DMEM)에 유지시켰다. 제조자 사용 설명에 따라서 Lipofectamine 2000 (Invitrogen)를 사용하여 0.8 x 105개의 HeLa 세포를 Cas9-암호화 플라스미드 (0.5ug) 및 sgRNA 발현 플라스미드 (0.5ug)로 형질감염시켰다.HeLa cells (ATCC, CCL-2) were incubated with 100 units / mL penicillin, 100 ug / mL streptomycin, 0.1 mM nonessential amino acids, and 10% (w / and maintained in Dulbecco's modified Eagle's medium (DMEM) supplemented with fetal bovine serum (FBS). 0.8 x 105 HeLa cells were transfected with Cas9-encoding plasmid (0.5 ug) and sgRNA expression plasmid (0.5 ug) using Lipofectamine 2000 (Invitrogen) according to the manufacturer's instructions.
실시예 3: Targeted deep sequencingExample 3: Targeted deep sequencing
NGS 라이브러리 구축을 위하여, Phusion polymerase (Thermo Fisher Scientific)를 사용하여 온-타겟 및 오프-타겟 영역 (표 1, 도 1b, 2b, 및 2d 참조)을 PCR 증폭시켰다. MiniSeq with TruSeq HT Dual Index system (Illumina)을 사용하여 제조자 사용 설명에 따라서 Pooled PCR amplicons을 시퀀싱하였다. Indel 빈도는 Cas-Analyzer(Park, J., Lim, K., Kim, J.S. & Bae, S. Cas-analyzer: an online tool for assessing genome editing results using NGS data.Bioinformatics33, 286-288 (2017))를 사용하여 측정하였다.For NGS library construction, on-target and off-target regions (see Table 1, Figures 1b, 2b, and 2d) were PCR amplified using Phusion polymerase (Thermo Fisher Scientific). Pooled PCR amplicons were sequenced using MiniSeq with TruSeq HT Dual Index system (Illumina) according to manufacturer's instructions. Indel frequency was measured using Cas-Analyzer (Park, J., Lim, K., Kim, JS & Bae, S. Cas-analyzer: an online tool for assessing genomic editing results using NGS data.Bioinformatics33 , 286-288 (2017) ).
실시예Example 4: 4:미스매칭Mismatching 5' 말단 5 'terminus뉴클레오타이드를Nucleotides 갖는 가이드 RNA의 유전자 교정 효율 시험 Genetic corrective efficiency test of guide RNA
진핵 세포에서 sgRNA를 발현하는데 일반적으로 사용되는 U6 프로모터는 전사를 개시하기 위하여 구아노신(G) 뉴클레오타이드를 필요로 하기 때문에 sgRNA는 전형적으로 5' 말단에 G 뉴클레오타이드를 포함한다. 대부분의 (평균 75%) DNA 표적 부위는 이 위치에서 미스매치를 포함하므로, 이들 부위에서의 gX19 sgRNA (도 1a; 도 1a에서 "g"는 미스매칭된 구아노신을, "G"는 매칭된 구아노신을 각각 나타내고, 각각의 N 또는 X는 A, T, G, 및 C를 포함하는 핵산 염기들 중에서 독립적으로 선택됨)와의 복합체화된 감쇠(attenuated) Cas9 변이체에 의한 세포 내 교정 수준은 낮아질 수 있다.The sgRNA typically contains a G nucleotide at the 5 'end because the U6 promoter commonly used to express sgRNA in eukaryotic cells requires a guanosine (G) nucleotide to initiate transcription. Most (average 75%) DNA target sites is because they contain a mismatch at this position, gX19 sgRNA (Fig. 1a in these regions; in Fig. 1a "g" is the mismatch of guano the shoes, "G" is matched The degree of intracellular correction by the attenuated Cas9 variant complexed with each of the N or X is independently selected from nucleic acid bases including A, T, G, and C can be lowered .
이와 같은 가설을 입증하기 위하여, gX19 sgRNA를 사용하여, HeLa 세포에서 5' 말단 뉴클레오타이드가 구아노신이 아닌 5개 유전자 부위에서의 eCas9-1.1과 Cas9-HF1의 유전자 교정 활성을 야생형 Cas9 단백질 (Cas9-WT)과 비교하여 그 결과를 도 1b에 나타내었다. 유전자 교정 활성은 실시예 3에 기재된 방법을 참조하여 idel 빈도를 측정하여 확인하였다.In order to prove the hypothesis, by using the gX19 sgRNA, the five eCas9-1.1 and gene correction activity of Cas9-HF1 in HeLa cells in the gene region 5 'terminal nucleotide is not a god guano Cas9 wild type protein (Cas9- WT). The results are shown in Fig. 1B. The genetic corrective activity was confirmed by measuring the idel frequency with reference to the method described in Example 3.
도 1b에 나타난 바와 같이, Cas9-WT는 5' 말단의 뉴클레오타이드 미스매치에 민감하지 않아서 48% 내지 78% (평균 70±5%)의 높은 빈도로 indels을 유도했다. eCas9-1.1은 5 개의 사이트 중 4 개의 사이트 (33±14%)에서 1.4~45% (22±10%)의 매우 낮은 indel 빈도를 보였다. Cas9-HF1은 3 개의 Cas9 뉴클레아제 중에서 가장 활성이 낮았으며, 빈도는 0.2 내지 20% (8.3±4.1%)의 indel 빈도를 보였다. CCR5 표적 부위에서, 2개의 감쇠된 Cas9 단백질은 모두 비활성이었다 (1% 미만의 indel 빈도를 보임). 또한 5' 말단에 추가의 구아노신을 갖는 gX20 sgRNA를 사용하여 유전자 교정 활성 (indel 빈도)을 시험하였다. 이들 sgRNA는 5' 말단에 매칭된 뉴클레오타이드를 갖는다. gX20 sgRNA는 AAVS1 및 HBB-02 부위에서 Cas9 변이체의 유전자 교정 활성을 증가시켰지만, 다른 세 부위에서는 gX19 sgRNA와 비교하여 Cas9 변이체의 유전자 교정 활성을 감소시켰다.As shown in FIG. 1 b, Cas9-WT was not sensitive to nucleotide mismatch at the 5 'terminus and induced indels with a high frequency of 48% to 78% (mean 70 ± 5%). eCas9-1.1 showed a very low indel frequency of 1.4 to 45% (22 ± 10%) at four of the five sites (33 ± 14%). Cas9-HF1 was the least active among the three Cas9 nuclease, and the frequency of indent was 0.2 to 20% (8.3 ± 4.1%). At the CCR5 target site, the two attenuated Cas9 proteins were all inactive (less than 1% indel frequency). In addition, gene corrective activity (indel frequency) was tested using gX20 sgRNA with additional guanosine at the 5 'end. These sgRNAs have nucleotides matched to the 5 ' end. gX20 sgRNA increased the gene correction activity of Cas9 mutants at AAVS1 and HBB-02 sites, but decreased the gene correction activity of Cas9 mutants compared to gX19 sgRNA at the other three sites.
실시예Example 5: 매칭된 5' 5: Matching 5 '뉴클레오타이드를Nucleotides 포함하는 IncludedsgRNA의sgRNA 제작 및 이를 이용한 Cas9 변이체의 유전자 교정 효율 시험 Genetic testing of Cas9 variants
높은 충실도의 Cas9 변이체로 타겟팅할 수 있는 유전자 부위의 수를 확대하기 위하여, 자가 절단 리보자임(self-cleaving ribozyme)을 이용하여 5' 뉴클레오타이드가 표적 DNA 서열과 매칭되는 sgRNA를 생산하였다. 각각의 sgRNA는 5' 말단에서 Hammerhead (HH) 리보자임에 융합되어 (Gao, Y. & Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing.Journal of integrative plant biology56, 343-349 (2014)), 자가 절단 후 성숙한 20-뉴클레오타이드 (X20) sgRNAs를 생성하였다. 상기 과정을 도 2a에 모식적으로 나타내었다.To amplify the number of targetable gene regions with high fidelity Cas9 mutants, sgRNAs were generated in which 5 'nucleotides matched the target DNA sequence using self-cleaving ribozyme. Each sgRNA is fused to the Hammerhead (HH) ribozyme at the 5 'end (Gao, Y. & Zhao, Y. Self-processing of ribosyl-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing.Journal of integrative plant biology 56, 343-349 (2014)), and then was cut self-generate the mature 20-nucleotide (X20) sgRNAs. The above process is schematically shown in Fig.
매칭된 5' 뉴클레오타이드를 갖는 HH 리보자임-융합 sgRNA (HH-X20으로 명명) 또는 미스매치 5' 구아노신 뉴클레오타이드를 갖는 HH 리보자임-융합 sgRNA (HH-gX19로 명명)를 Cas9-WT 또는 높은 충실도의 Cas9 변이체와 조합하여 HeLa 세포에서의 유전자 교정 활성을 시험하여, 그 결과를 도 2b 및 도 2c에 나타내었다. 유전자 교정 활성은 실시예 3에 기재된 방법을 참조하여 idel 빈도를 측정하여 확인하였다.HH ribozyme-fused sgRNA (designated HH-gX19 ) with a matched 5 'nucleotide and a mature mature sgRNA (named HH-X20 ) or a mismatch 5' guanosine nucleotide with Cas9-WT or The gene correcting activity in HeLa cells was tested in combination with Cas9 variants with high fidelity, and the results are shown in FIGS. 2B and 2C. The genetic corrective activity was confirmed by measuring the idel frequency with reference to the method described in Example 3.
도 2b에 나타난 바와 같이, HH-X20 sgRNA를 사용함으로써 시험된 2 개의 Cas9 변이체 (eCas9-1.1 및 Cas9-HF1) 모두 5 개의 모든 표적 부위에서 교정 활성을 나타낼 수 있게 되었다. HH-X20 sgRNA와 함께 eCas9-1.1 (69±5%) 또는 Cas9-HF1 (59±7%)를 사용하여 얻은 indel 빈도는 HH-X20 또는 HH-gX19sgRNA와 함께 Cas9-WT (각각 71±6% 또는 70±3%)를 사용하여 얻은 결과와 거의 유사하게 나타났다 (도 2c). Cas9 변이체 eCas9-1.1 및 Cas9-HF1는 HH-gX19 sgRNA와 함께 사용되는 경우 낮은 교정 활성을 보였으며, 이러한 결과는 높은 충실도의 변이체는 리보자임과의 융합 그 자체 보다는 5' 말단에 매칭된 뉴클레오타이드의 존재 때문에 높은 유전자 교정 활성을 유지할 수 있음을 보여준다.As shown in Fig. 2b, both of the Cas9 variants tested (eCas9-1.1 and Cas9-HF1) by using HH-X20 sgRNA were able to show corrective activity at all five target sites. The indel level obtained using eCas9-1.1 (69 ± 5%) or Cas9-HF1 (59 ± 7%) with HH-X20 sgRNA was measured using HH-X20 or HH-gX19 (Fig. 2c), using Cas9-WT (71 ± 6% or 70 ± 3%, respectively) with sgRNA. Cas9 variant eCas9-1.1 and Cas9-HF1 is showed low activity when used with the correction HH-gX19 sgRNA, these results are variants of high fidelity matching nucleotides at the 5 'terminus than the fusion itself and the ribozyme Lt; RTI ID = 0.0 > genetic < / RTI > correction activity.
다음으로, HeLa 세포에서 알려진 오프-타겟 위치에서의 돌연변이 빈도를 측정하여, HH-X20 sgRNAs와 함께 사용되는 경우의 2 가지 Cas9 변이체의 표적 특이도를 비교하였다 (CCR5 특이적 sgRNA에 대해서는 오프-타겟 위치가 알려지지 않아서 분석에서 제외함). 상기 얻어진 결과를 도 2d에 나타내었다. 도 2d에 나타난 바와 같이, 각각의 on-target site와 1 내지 3 개의 뉴클레오타이드가 상이한 off-target site의 대부분에서, 2 종의 Cas9 변이체 모두 Cas9-WT보다 낮은 indel 빈도를 나타냈다. Cas9-HF1은 하나의 뉴클레오타이드 미스매치를 갖는 3 개의 오프-타겟 사이트 (하나의 HBB-03 오프-타겟 사이트 및 2 개의 HBB-04 오프-타겟 사이트)를 구별할 수 있었다.Next, the mutation frequencies at known off-target positions in HeLa cells were measured to compare the target specificity of the two Cas9 variants when used with HH-X20 sgRNAs (for off-target sites for CCR5-specific sgRNA, Excluded from analysis because target location is unknown.) The results obtained are shown in Fig. 2d. As shown in Fig. 2d, both of the Cas9 mutants showed lower indel frequencies than Cas9-WT in most of the off-target sites where one or three nucleotides differ from each on-target site. Cas9-HF1 was able to distinguish three off-target sites (one HBB-03 off-target site and two HBB-04 off-target sites) with one nucleotide mismatch.
상기 결과를 요약하면, 야생형 단백질과 달리, 새롭게 개발된 고특이성 Cas9 변이체가 5' 말단에서 미스매칭을 갖는 표적 부위에서 교정 효율이 낮은 경우가 많음을 보여주며, 이러한 결과는 5' 뉴클레오티드가 원핵 세포 또는 인간 세포와 같은 진핵 세포에서 CRISPR-Cas9의 높은 특이성에 기여함을 최초로 보여주는 것이다. HH-리보자임 융합의 자기 분해 활성에 의하여 sgRNA의 첫 번째 뉴클레오타이드를 표적 DNA 서열과 매칭시킴으로써, 온-타겟 교정 효율에 불리한 영향 없이 유전체 교정의 높은 특이성을 달성할 수 있다. HH-리보자임 융합을 사용하는 것에 대한 대안으로서, tRNA 융합(Port, F. & Bullock, S.L. Augmenting CRISPR applications in Drosophila with tRNA-flanked sgRNAs.Nature methods13, 852-854 (2016))또는 화학적 합성 (Hendel, A. et al. Chemically modified guide RNAs enhance CRISPR-Cas genome editing in human primary cells.Nature biotechnology33, 985-989 (2015))을 통해 5' 말단에 G 이외의 매칭된 뉴클레오타이드 (5' non-G nucleotide)를 갖는 sgRNA를 제작하고 상기한 2 종의 high-fidelity Cas9 변이체와 조합하여 사용될 수 있다. Cas9- 및 sgRNA-암호화 플라스미드를 사용하는 경우와 비교하여, 사전 조립된 (생체 외 또는 세포 외에서 조립(제작)된) Cas9 변이체 리보핵산단백질(Kim, S., Kim, D., Cho, S.W., Kim, J. & Kim, J.S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins.Genome research (2014))의 전달에 의하여 genome-wide target specificity를 보다 향상시킬 수 있다.In summary, the results show that, in contrast to wild-type proteins, the newly developed high specificity Cas9 mutants are often less efficient at the target sites with mismatch at the 5 'end, and these results indicate that the 5' Or to the high specificity of CRISPR-Cas9 in eukaryotic cells such as human cells. By matching the first nucleotide of the sgRNA with the target DNA sequence by the autolytic activity of the HH-ribozyme fusion, high specificity of the genetic modification can be achieved without adversely affecting the on-target calibration efficiency. As an alternative to using HH-ribozyme fusion, tRNA fusion (Port, F. & Bullock, SL Augmenting CRISPR applications in Drosophila with tRNA-flanked sgRNAs.Nature methods13 , 852-854 (2016) Or chemically synthesized (Hendel, A. et al., Chemically modified guide RNAs enhance CRISPR-Cas genome editing in human primary cells,Nature Biotechnology33 , 985-989 (2015) 5 'non-G nucleotide) and can be used in combination with the above two kinds of high-fidelity Cas9 mutants. Kim, S., Kim, D., Cho, SW, et < RTI ID = 0.0 > al., ≪ / RTI >Genome -wide target specificity can be further improved by the delivery of Cas9 ribonucleoproteins.Genome research (2014). Kim, J. & Kim, JS Highly efficient RNA-guided genome editing.
<110> INSTITUTE FOR BASIC SCIENCE<120> Gene editing composition comprising sgRNAs with matched 5' nucleotide and gene editing method using the same<130> DPP20181455KR<150> KR 10-2017-0064332<151> 2017-05-24<160> 12<170> KopatentIn 3.0<210> 1<211> 12<212> RNA<213> Artificial Sequence<220><223> Essential part of crRNA<400> 1guuuuagagc ua 12<210> 2<211> 10<212> RNA<213> Artificial Sequence<220><223> 3'-terminal part of crRNA<400> 2ugcuguuuug 10<210> 3<211> 60<212> RNA<213> Artificial Sequence<220><223> Essential part of tracrRNA<400> 3uagcaaguua aaauaaggcu aguccguuau caacuugaaa aaguggcacc gagucggugc 60 60<210> 4<211> 1368<212> PRT<213> Artificial Sequence<220><223> Cas9 from Streptococcus pyogenes<400> 4Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val 1 5 10 15 Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe 20 25 30 Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile 35 40 45 Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu 50 55 60 Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys 65 70 75 80 Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser 85 90 95 Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys 100 105 110 His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr 115 120 125 His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp 130 135 140 Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His145 150 155 160 Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro 165 170 175 Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr 180 185 190 Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala 195 200 205 Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn 210 215 220 Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn225 230 235 240 Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe 245 250 255 Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp 260 265 270 Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp 275 280 285 Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp 290 295 300 Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser305 310 315 320 Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys 325 330 335 Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe 340 345 350 Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser 355 360 365 Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp 370 375 380 Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg385 390 395 400 Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu 405 410 415 Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe 420 425 430 Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile 435 440 445 Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp 450 455 460 Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu465 470 475 480 Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr 485 490 495 Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser 500 505 510 Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys 515 520 525 Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln 530 535 540 Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr545 550 555 560 Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp 565 570 575 Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly 580 585 590 Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp 595 600 605 Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr 610 615 620 Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala625 630 635 640 His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr 645 650 655 Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp 660 665 670 Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe 675 680 685 Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe 690 695 700 Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu705 710 715 720 His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly 725 730 735 Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly 740 745 750 Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln 755 760 765 Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile 770 775 780 Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro785 790 795 800 Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu 805 810 815 Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg 820 825 830 Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys 835 840 845 Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg 850 855 860 Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys865 870 875 880 Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys 885 890 895 Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp 900 905 910 Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr 915 920 925 Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp 930 935 940 Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser945 950 955 960 Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg 965 970 975 Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val 980 985 990 Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe 995 1000 1005 Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala Lys 1010 1015 1020 Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser1025 1030 1035 1040 Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu 1045 1050 1055 Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu Ile 1060 1065 1070 Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val Leu Ser 1075 1080 1085 Met Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln Thr Gly Gly 1090 1095 1100 Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile1105 1110 1115 1120 Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp Ser 1125 1130 1135 Pro Thr Val Ala Tyr Ser Val Leu Val Val Ala Lys Val Glu Lys Gly 1140 1145 1150 Lys Ser Lys Lys Leu Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile 1155 1160 1165 Met Glu Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala 1170 1175 1180 Lys Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys1185 1190 1195 1200 Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser 1205 1210 1215 Ala Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr 1220 1225 1230 Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser 1235 1240 1245 Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys His 1250 1255 1260 Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val1265 1270 1275 1280 Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys 1285 1290 1295 His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His Leu 1300 1305 1310 Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr Phe Asp 1315 1320 1325 Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu Val Leu Asp 1330 1335 1340 Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile1345 1350 1355 1360 Asp Leu Ser Gln Leu Gly Gly Asp 1365 <210> 5<211> 4107<212> DNA<213> Artificial Sequence<220><223> Cas9-coding sequence<400> 5atggacaaga agtacagcat cggcctggac atcggtacca acagcgtggg ctgggccgtg 60atcaccgacg agtacaaggt gcccagcaag aagttcaagg tgctgggcaa caccgaccgc 120cacagcatca agaagaacct gatcggcgcc ctgctgttcg acagcggcga gaccgccgag 180gccacccgcc tgaagcgcac cgcccgccgc cgctacaccc gccgcaagaa ccgcatctgc 240tacctgcagg agatcttcag caacgagatg gccaaggtgg acgacagctt cttccaccgc 300ctggaggaga gcttcctggt ggaggaggac aagaagcacg agcgccaccc catcttcggc 360aacatcgtgg acgaggtggc ctaccacgag aagtacccca ccatctacca cctgcgcaag 420aagctggtgg acagcaccga caaggccgac ctgcgcctga tctacctggc cctggcccac 480atgatcaagt tccgcggcca cttcctgatc gagggcgacc tgaaccccga caacagcgac 540gtggacaagc tgttcatcca gctggtgcag acctacaacc agctgttcga ggagaacccc 600atcaacgcca gcggcgtgga cgccaaggcc atcctgagcg cccgcctgag caagagccgc 660cgcctggaga acctgatcgc ccagctgccc ggcgagaaga agaacggcct gttcggcaac 720ctgatcgccc tgagcctggg cctgaccccc aacttcaaga gcaacttcga cctggccgag 780gacgccaagc tgcagctgag caaggacacc tacgacgacg acctggacaa cctgctggcc 840cagatcggcg accagtacgc cgacctgttc ctggccgcca agaacctgag cgacgccatc 900ctgctgagcg acatcctgcg cgtgaacacc gagatcacca aggcccccct gagcgccagc 960atgatcaagc gctacgacga gcaccaccag gacctgaccc tgctgaaggc cctggtgcgc 1020cagcagctgc ccgagaagta caaggagatc ttcttcgacc agagcaagaa cggctacgcc 1080ggctacatcg acggcggcgc cagccaggag gagttctaca agttcatcaa gcccatcctg 1140gagaagatgg acggcaccga ggagctgctg gtgaagctga accgcgagga cctgctgcgc 1200aagcagcgca ccttcgacaa cggcagcatc ccccaccaga tccacctggg cgagctgcac 1260gccatcctgc gccgccagga ggacttctac cccttcctga aggacaaccg cgagaagatc 1320gagaagatcc tgaccttccg catcccctac tacgtgggcc ccctggcccg cggcaacagc 1380cgcttcgcct ggatgacccg caagagcgag gagaccatca ccccctggaa cttcgaggag 1440gtggtggaca agggcgccag cgcccagagc ttcatcgagc gcatgaccaa cttcgacaag 1500aacctgccca acgagaaggt gctgcccaag cacagcctgc tgtacgagta cttcaccgtg 1560tacaacgagc tgaccaaggt gaagtacgtg accgagggca tgcgcaagcc cgccttcctg 1620agcggcgagc agaagaaggc catcgtggac ctgctgttca agaccaaccg caaggtgacc 1680gtgaagcagc tgaaggagga ctacttcaag aagatcgagt gcttcgacag cgtggagatc 1740agcggcgtgg aggaccgctt caacgccagc ctgggcacct accacgacct gctgaagatc 1800atcaaggaca aggacttcct ggacaacgag gagaacgagg acatcctgga ggacatcgtg 1860ctgaccctga ccctgttcga ggaccgcgag atgatcgagg agcgcctgaa gacctacgcc 1920cacctgttcg acgacaaggt gatgaagcag ctgaagcgcc gccgctacac cggctggggc 1980cgcctgagcc gcaagcttat caacggcatc cgcgacaagc agagcggcaa gaccatcctg 2040gacttcctga agagcgacgg cttcgccaac cgcaacttca tgcagctgat ccacgacgac 2100agcctgacct tcaaggagga catccagaag gcccaggtga gcggccaggg cgacagcctg 2160cacgagcaca tcgccaacct ggccggcagc cccgccatca agaagggcat cctgcagacc 2220gtgaaggtgg tggacgagct ggtgaaggtg atgggccgcc acaagcccga gaacatcgtg 2280atcgagatgg cccgcgagaa ccagaccacc cagaagggcc agaagaacag ccgcgagcgc 2340atgaagcgca tcgaggaggg catcaaggag ctgggcagcc agatcctgaa ggagcacccc 2400gtggagaaca cccagctgca gaacgagaag ctgtacctgt actacctgca gaacggccgc 2460gacatgtacg tggaccagga gctggacatc aaccgcctga gcgactacga cgtggaccac 2520atcgtgcccc agagcttcct gaaggacgac agcatcgaca acaaggtgct gacccgcagc 2580gacaagaacc gcggcaagag cgacaacgtg cccagcgagg aggtggtgaa gaagatgaag 2640aactactggc gccagctgct gaacgccaag ctgatcaccc agcgcaagtt cgacaacctg 2700accaaggccg agcgcggcgg cctgagcgag ctggacaagg ccggcttcat caagcgccag 2760ctggtggaga cccgccagat caccaagcac gtggcccaga tcctggacag ccgcatgaac 2820accaagtacg acgagaacga caagctgatc cgcgaggtga aggtgatcac cctgaagagc 2880aagctggtga gcgacttccg caaggacttc cagttctaca aggtgcgcga gatcaacaac 2940taccaccacg cccacgacgc ctacctgaac gccgtggtgg gcaccgccct gatcaagaag 3000taccccaagc tggagagcga gttcgtgtac ggcgactaca aggtgtacga cgtgcgcaag 3060atgatcgcca agagcgagca ggagatcggc aaggccaccg ccaagtactt cttctacagc 3120aacatcatga acttcttcaa gaccgagatc accctggcca acggcgagat ccgcaagcgc 3180cccctgatcg agaccaacgg cgagaccggc gagatcgtgt gggacaaggg ccgcgacttc 3240gccaccgtgc gcaaggtgct gagcatgccc caggtgaaca tcgtgaagaa gaccgaggtg 3300cagaccggcg gcttcagcaa ggagagcatc ctgcccaagc gcaacagcga caagctgatc 3360gcccgcaaga aggactggga ccccaagaag tacggcggct tcgacagccc caccgtggcc 3420tacagcgtgc tggtggtggc caaggtggag aagggcaaga gcaagaagct gaagagcgtg 3480aaggagctgc tgggcatcac catcatggag cgcagcagct tcgagaagaa ccccatcgac 3540ttcctggagg ccaagggcta caaggaggtg aagaaggacc tgatcatcaa gctgcccaag 3600tacagcctgt tcgagctgga gaacggccgc aagcgcatgc tggccagcgc cggcgagctg 3660cagaagggca acgagctggc cctgcccagc aagtacgtga acttcctgta cctggccagc 3720cactacgaga agctgaaggg cagccccgag gacaacgagc agaagcagct gttcgtggag 3780cagcacaagc actacctgga cgagatcatc gagcagatca gcgagttcag caagcgcgtg 3840atcctggccg acgccaacct ggacaaggtg ctgagcgcct acaacaagca ccgcgacaag 3900cccatccgcg agcaggccga gaacatcatc cacctgttca ccctgaccaa cctgggcgcc 3960cccgccgcct tcaagtactt cgacaccacc atcgaccgca agcgctacac cagcaccaag 4020gaggtgctgg acgccaccct gatccaccag agcatcaccg gtctgtacga gacccgcatc 4080gacctgagcc agctgggcgg cgactaa 4107<210> 6<211> 21<212> DNA<213> Artificial Sequence<220><223> NLS<400> 6cccaagaaga agaggaaagt c 21<210> 7<211> 23<212> DNA<213> Artificial Sequence<220><223> Target sequence on AAVS1 and PAM<400> 7ctccctccca ggatcctctc tgg 23<210> 8<211> 23<212> DNA<213> Artificial Sequence<220><223> Target sequence on CCR5 and PAM<400> 8tcatcctgat aaactgcaaa agg 23<210> 9<211> 23<212> DNA<213> Artificial Sequence<220><223> Target sequence on HBB-02 and PAM<400> 9cttgccccac agggcagtaa cgg 23<210> 10<211> 23<212> DNA<213> Artificial Sequence<220><223> Target sequence on HBB-03 and PAM<400> 10cacgttcacc ttgccccaca ggg 23<210> 11<211> 23<212> DNA<213> Artificial Sequence<220><223> Target sequence on HBB-04 and PAM<400> 11ccacgttcac cttgccccac agg 23<210> 12<211> 23<212> DNA<213> Artificial Sequence<220><223> Target sequence on EMX1-05 and PAM<400> 12tgtactttgt cctccggttg tgg 23<110> INSTITUTE FOR BASIC SCIENCE<120> Gene editing composition comprising sgRNAs with matched 5 ' nucleotide and gene editing method using the same<130> DPP20181455KR<150> KR 10-2017-0064332<151> 2017-05-24<160> 12<170> Kopatentin 3.0<210> 1<211> 12<212> RNA<213> Artificial Sequence<220><223> Essential part of crRNA<400> 1guuuuagagc ua 12<210> 2<211> 10<212> RNA<213> Artificial Sequence<220>The 3'-terminal part of the crRNA<400> 2ugcuguuuug 10<210> 3<211> 60<212> RNA<213> Artificial Sequence<220><223> Essential part of tracrRNA<400> 3uagcaaguua aaauaaggcu aguccguuau caacuugaaa aaguggcacc gagucggugc 60 60<210> 4<211> 1368<212> PRT<213> Artificial Sequence<220><223> Cas9 from Streptococcus pyogenes<400> 4Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val 1 5 10 15Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe 20 25 30Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile 35 40 45Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu 50 55 60Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys 65 70 75 80Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser 85 90 95Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys 100 105 110His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr 115 120 125His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp 130 135 140Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His145 150 155 160Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro 165 170 175Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr 180 185 190Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala 195 200 205Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn 210 215 220Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn225 230 235 240Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe 245 250 255Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp 260 265 270Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp 275 280 285Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp 290 295 300Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser305 310 315 320Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys 325 330 335Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe 340 345 350Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser 355 360 365Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp 370 375 380Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg385 390 395 400Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu 405 410 415Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe 420 425 430Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile 435 440 445Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp 450 455 460Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu465 470 475 480Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr 485 490 495Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser 500 505 510Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys 515 520 525Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln 530 535 540Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr545 550 555 560Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp 565 570 575Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly 580 585 590Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp 595 600 605Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr 610 615 620Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala625 630 635 640His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr 645 650 655Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp 660 665 670Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe 675 680 685Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe 690 695 700Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu705 710 715 720His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly 725 730 735Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly 740 745 750Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln 755 760 765Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile 770 775 780Glu Glu Ile Lys Glu Leu Gly Ser Glu Ile Leu Lys Glu His Pro785 790 795 800Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu 805 810 815Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg 820 825 830Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys 835 840 845Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg 850 855 860Gly Lys Ser Asp Val Val Ser Ser Glu Val Val Lys Lys Met Lys865 870 875 880Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys 885 890 895Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp 900 905 910Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr 915 920 925Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp 930 935 940Glu Asn Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser945 950 955 960Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg 965 970 975Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val 980 985 990Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe 995 1000 1005Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala Lys 1010 1015 1020Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser1025 1030 1035 1040Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu 1045 1050 1055Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu Ile 1060 1065 1070Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val Leu Ser 1075 1080 1085Met Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln Thr Gly Gly 1090 1095 1100Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile1105 1110 1115 1120Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp Ser 1125 1130 1135Pro Thr Val Ala Tyr Ser Val Leu Val Val Ala Lys Val Glu Lys Gly 1140 1145 1150Lys Ser Lys Lys Leu Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile 1155 1160 1165Met Glu Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala 1170 1175 1180Lys Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys1185 1190 1195 1200Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser 1205 1210 1215Ala Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr 1220 1225 1230Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser 1235 1240 1245Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys His 1250 1255 1260Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val1265 1270 1275 1280Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys 1285 1290 1295His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His Leu 1300 1305 1310Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr Phe Asp 1315 1320 1325Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu Val Leu Asp 1330 1335 1340Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile1345 1350 1355 1360Asp Leu Ser Gln Leu Gly Gly Asp 1365<210> 5<211> 4107<212> DNA<213> Artificial Sequence<220><223> Cas9-coding sequence<400> 5atggacaaga agtacagcat cggcctggac atcggtacca acagcgtggg ctgggccgtg 60atcaccgacg agtacaaggt gcccagcaag aagttcaagg tgctgggcaa caccgaccgc 120cacagcatca agaagaacct gatcggcgcc ctgctgttcg acagcggcga gaccgccgag 180gccacccgcc tgaagcgcac cgcccgccgc cgctacaccc gccgcaagaa ccgcatctgc 240tacctgcagg agatcttcag caacgagatg gccaaggtgg acgacagctt cttccaccgc 300ctggaggaga gcttcctggt ggaggaggac aagaagcacg agcgccaccc catcttcggc 360aacatcgtgg acgaggtggc ctaccacgag aagtacccca ccatctacca cctgcgcaag 420cctggcccacatgatcaagt tccgcggcca cttcctgatc gagggcgacc tgaaccccga caacagcgac 540gtggacaagc tgttcatcca gctggtgcag acctacaacc agctgttcga ggagaacccc 600atcaacgcca gcggcgtgga cgccaaggcc atcctgagcg cccgcctgag caagagccgc 660cgcctggaga acctgatcgc ccagctgccc ggcgagaaga agaacggcct gttcggcaac 720ctgatcgccc tgagcctggg cctgaccccc aacttcaaga gcaacttcga cctggccgag 780gacgccaagc tgcagctgag caaggacacc tacgacgacg acctggacaa cctgctggcc 840cagatcggcg accagtacgc cgacctgttc ctggccgcca agaacctgag cgacgccatc 900ctgctgagcg acatcctgcg cgtgaacacc gagatcacca aggcccccct gagcgccagc 960atgatcaagc gctacgacga gcaccaccag gacctgaccc tgctgaaggc cctggtgcgc 1020cagcagctgc ccgagaagta caaggagatc ttcttcgacc agagcaagaa cggctacgcc 1080ggctacatcg acggcggcgc cagccaggag gagttctaca agttcatcaa gcccatcctg 1140gagaagatgg acggcaccga ggagctgctg gtgaagctga accgcgagga cctgctgcgc 1200aagcagcgca ccttcgacaa cggcagcatc ccccaccaga tccacctggg cgagctgcac 1260gccatcctgc gccgccagga ggacttctac cccttcctga aggacaaccg cgagaagatc 1320gagaagatcc tgaccttccg catcccctac tacgtgggcc ccctggcccg cggcaacagc 1380cgcttcgcct ggatgacccg caagagcgag gagaccatca ccccctggaa cttcgaggag 1440gtggtggaca agggcgccag cgcccagagc ttcatcgagc gcatgaccaa cttcgacaag 1500aacctgccca acgagaaggt gctgcccaag cacagcctgc tgtacgagta cttcaccgtg 1560tacaacgagc tgaccaaggt gaagtacgtg accgagggca tgcgcaagcc cgccttcctg 1620agcggcgagc agaagaaggc catcgtggac ctgctgttca agaccaaccg caaggtgacc 1680gtgaagcagc tgaaggagga ctacttcaag aagatcgagt gcttcgacag cgtggagatc 1740agcggcgtgg aggaccgctt caacgccagc ctgggcacct accacgacct gctgaagatc 1800atcaaggaca aggacttcct ggacaacgag gagaacgagg acatcctgga ggacatcgtg 1860ctgaccctga ccctgttcga ggaccgcgag atgatcgagg agcgcctgaa gacctacgcc 1920cacctgttcg acgacaaggt gatgaagcag ctgaagcgcc gccgctacac cggctggggc 1980cgcctgagcc gcaagcttat caacggcatc cgcgacaagc agagcggcaa gaccatcctg 2040gacttcctga agagcgacgg cttcgccaac cgcaacttca tgcagctgat ccacgacgac 2100agcctgacct tcaaggagga catccagaag gcccaggtga gcggccaggg cgacagcctg 2160ccgagcaca tcgccaacct ggccggcagc cccgccatca agaagggcat cctgcagacc 2220gtgaaggtgg tggacgagct ggtgaaggtg atgggccgcc acaagcccga gaacatcgtg 2280atcgagatgg cccgcgagaa ccagaccacc cagaagggcc agaagaacag ccgcgagcgc 2340atgaagcgca tcgaggaggg catcaaggag ctgggcagcc agatcctgaa ggagcacccc 2400gtggagaaca cccagctgca gaacgagaag ctgtacctgt actacctgca gaacggccgc 2460gacatgtacg tggaccagga gctggacatc aaccgcctga gcgactacga cgtggaccac 2520atcgtgcccc agagcttcct gaaggacgac agcatcgaca acaaggtgct gacccgcagc 2580gacaagaacc gcggcaagag cgacaacgtg cccagcgagg aggtggtgaa gaagatgaag 2640aactactggc gccagctgct gaacgccaag ctgatcaccc agcgcaagtt cgacaacctg 2700accaaggccg agcgcggcgg cctgagcgag ctggacaagg ccggcttcat caagcgccag 2760ctggtggaga cccgccagat caccaagcac gtggcccaga tcctggacag ccgcatgaac 2820accaagtacg acgagaacga caagctgatc cgcgaggtga aggtgatcac cctgaagagc 2880aagctggtga gcgacttccg caaggacttc cagttctaca aggtgcgcga gatcaacaac 2940taccaccacg cccacgacgc ctacctgaac gccgtggtgg gcaccgccct gatcaagaag 3000taccccaagc tggagagcga gttcgtgtac ggcgactaca aggtgtacga cgtgcgcaag 3060atgatcgcca agagcgagca ggagatcggc aaggccaccg ccaagtactt cttctacagc 3120aacatcatga acttcttcaa gaccgagatc accctggcca acggcgagat ccgcaagcgc 3180cccctgatcg agaccaacgg cgagaccggc gagatcgtgt gggacaaggg ccgcgacttc 3240gccaccgtgc gcaaggtgct gagcatgccc caggtgaaca tcgtgaagaa gaccgaggtg 3300cagaccggcg gcttcagcaa ggagagcatc ctgcccaagc gcaacagcga caagctgatc 3360gcccgcaaga aggactggga ccccaagaag tacggcggct tcgacagccc caccgtggcc 3420tacagcgtgc tggtggtggc caaggtggag aagggcaaga gcaagaagct gaagagcgtg 3480aaggagctgc tgggcatcac catcatggag cgcagcagct tcgagaagaa ccccatcgac 3540ttcctggagg ccaagggcta caaggaggtg aagaaggacc tgatcatcaa gctgcccaag 3600tacagcctgt tcgagctgga gaacggccgc aagcgcatgc tggccagcgc cggcgagctg 3660cagaagggca acgagctggc cctgcccagc aagtacgtga acttcctgta cctggccagc 3720cactacgaga agctgaaggg cagccccgag gacaacgagc agaagcagct gttcgtggag 3780cagcacaagc actacctgga cagagatcatc gagcagatca gcgagttcag caagcgcgtg 3840atcctggccg acgccaacct ggacaaggtg ctgagcgcct acaacaagca ccgcgacaag 3900cccatccgcg agcaggccga gaacatcatc cacctgttca ccctgaccaa cctgggcgcc 3960cccgccgcct tcaagtactt cgacaccacc atcgaccgca agcgctacac cagcaccaag 4020gaggtgctgg acgccaccct gatccaccag agcatcaccg gtctgtacga gacccgcatc 4080gacctgagcc agctgggcgg cgactaa 4107<210> 6<211> 21<212> DNA<213> Artificial Sequence<220><223> NLS<400> 6cccaagaaga agaggaaagt c 21<210> 7<211> 23<212> DNA<213> Artificial Sequence<220><223> Target sequence on AAVS1 and PAM<400> 7ctccctccca ggatcctctc tgg 23<210> 8<211> 23<212> DNA<213> Artificial Sequence<220><223> Target sequence on CCR5 and PAM<400> 8tcatcctgat aaactgcaaa agg 23<210> 9<211> 23<212> DNA<213> Artificial Sequence<220><223> Target sequence on HBB-02 and PAM<400> 9cttgccccac agggcagtaa cgg 23<210> 10<211> 23<212> DNA<213> Artificial Sequence<220><223> Target sequence on HBB-03 and PAM<400> 10cacgttcacc ttgccccaca ggg 23<210> 11<211> 23<212> DNA<213> Artificial Sequence<220><223> Target sequence on HBB-04 and PAM<400> 11ccacgttcac cttgccccac agg 23<210> 12<211> 23<212> DNA<213> Artificial Sequence<220><223> Target sequence on EMX1-05 and PAM<400> 12tgtactttgt cctccggttg tgg 23
Claims (10)
Translated fromKorean상기 고특이성 Cas9 변이체는 Cas9 단백질의 알라닌이 아닌 하나 이상의 아미노산 잔기가 알라닌으로 치환된 것이고,
상기 매칭된 5' 뉴클레오타이드는 표적 서열의 5' 말단 뉴클레오타이드와 매칭되는 염기를 포함하는 뉴클레오타이드인,
유전자 교정용 조성물.A guide RNA comprising a high specificity Cas9 mutant and a matched 5 ' nucleotide,
Wherein said high specificity Cas9 mutant is one in which at least one amino acid residue which is not alanine of Cas9 protein is substituted with alanine,
Wherein the matched 5 ' nucleotide is a nucleotide comprising a base that matches the 5 ' terminal nucleotide of the target sequence,
Composition for gene correction.
5'-(Ncas9)l-(서열번호 1)-(올리고뉴클레오타이드 링커)-(서열번호 3)-3'
상기 서열 일반식에서, (Ncas9)l는 표적 서열과 혼성화 가능 표적화 서열이고, l은 상기 표적화 서열에 포함된 뉴클레오타이드 수로서 15 내지 30의 정수이고,
상기 올리고뉴클레오타이드 링커는 A, U, C 및 G로 이루어진 군에서 각각 독립적으로 선택된 3 내지 5개의 뉴클레오타이드를 포함함.2. The composition according to claim 1, wherein the guide RNA is sgRNA (single-guide RNA) represented by the following general formula:
5 '- (Ncas9 )1 - (SEQ ID NO: 1) - (oligonucleotide linker) - (SEQ ID NO: 3) -3'
(Ncas9 )1 is a hybridization target sequence with a target sequence, 1 is the number of nucleotides contained in the targeting sequence, and is an integer of 15 to 30,
Wherein the oligonucleotide linker comprises 3 to 5 nucleotides each independently selected from the group consisting of A, U, C and G.
(2) 상기 sgRNA의 5' 말단, 3' 말단, 또는 양 말단에 융합된 자가-절단 활성을 갖는 RNA 절단효소 또는 1 내지 6개의 tRNA를 포함하는, 융합 RNA 분자.(1) sgRNA and
(2) a fusion RNA molecule comprising an RNA-cleaving enzyme or one to six tRNAs having self-cleavage activity fused to the 5'-, 3'-, or both ends of the sgRNA.
Applications Claiming Priority (2)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| KR1020170064332 | 2017-05-24 | ||
| KR20170064332 | 2017-05-24 |
Publications (2)
| Publication Number | Publication Date |
|---|---|
| KR20180128864Atrue KR20180128864A (en) | 2018-12-04 |
| KR102151064B1 KR102151064B1 (en) | 2020-09-02 |
Family
ID=64669239
Family Applications (1)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| KR1020180059165AActiveKR102151064B1 (en) | 2017-05-24 | 2018-05-24 | Gene editing composition comprising sgRNAs with matched 5' nucleotide and gene editing method using the same |
Country Status (1)
| Country | Link |
|---|---|
| KR (1) | KR102151064B1 (en) |
Cited By (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN112680430A (en)* | 2020-12-28 | 2021-04-20 | 南方医科大学 | CRISPR SpCas9 mutant and application thereof |
| CN117866926A (en)* | 2024-03-07 | 2024-04-12 | 珠海舒桐医疗科技有限公司 | A CRISPR-FrCas9 protein mutant and its application |
| CN120519430A (en)* | 2025-07-25 | 2025-08-22 | 珠海舒桐医疗科技有限公司 | Compact CRISPR/Cas9 gene editing system and application |
Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2016073433A1 (en)* | 2014-11-06 | 2016-05-12 | E. I. Du Pont De Nemours And Company | Peptide-mediated delivery of rna-guided endonuclease into cells |
| WO2016205613A1 (en)* | 2015-06-18 | 2016-12-22 | The Broad Institute Inc. | Crispr enzyme mutations reducing off-target effects |
- 2018
- 2018-05-24KRKR1020180059165Apatent/KR102151064B1/enactiveActive
Patent Citations (2)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| WO2016073433A1 (en)* | 2014-11-06 | 2016-05-12 | E. I. Du Pont De Nemours And Company | Peptide-mediated delivery of rna-guided endonuclease into cells |
| WO2016205613A1 (en)* | 2015-06-18 | 2016-12-22 | The Broad Institute Inc. | Crispr enzyme mutations reducing off-target effects |
Cited By (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN112680430A (en)* | 2020-12-28 | 2021-04-20 | 南方医科大学 | CRISPR SpCas9 mutant and application thereof |
| CN112680430B (en)* | 2020-12-28 | 2023-06-06 | 南方医科大学 | CRISPR SpCas9 mutant and application thereof |
| CN117866926A (en)* | 2024-03-07 | 2024-04-12 | 珠海舒桐医疗科技有限公司 | A CRISPR-FrCas9 protein mutant and its application |
| CN120519430A (en)* | 2025-07-25 | 2025-08-22 | 珠海舒桐医疗科技有限公司 | Compact CRISPR/Cas9 gene editing system and application |
Also Published As
| Publication number | Publication date |
|---|---|
| KR102151064B1 (en) | 2020-09-02 |
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| AU2021231074C1 (en) | Class II, type V CRISPR systems | |
| JP7038079B2 (en) | CRISPR hybrid DNA / RNA polynucleotide and usage | |
| CN107012164B (en) | CRISPR/Cpf1 plant genome directed modification functional unit, vector containing functional unit and application of functional unit | |
| CN111373041B (en) | CRISPR/CAS systems and methods for genome editing and transcription regulation | |
| KR102728416B1 (en) | Compositions and methods for gene editing for hemophilia A | |
| KR101906491B1 (en) | Composition for Genome Editing comprising Cas9 derived from F. novicida | |
| KR101817482B1 (en) | Genome editing using campylobacter jejuni crispr/cas system-derived rgen | |
| KR20180069898A (en) | Nucleobase editing agents and uses thereof | |
| KR102151065B1 (en) | Composition and method for base editing in animal embryos | |
| CA3009727A1 (en) | Compositions and methods for the treatment of hemoglobinopathies | |
| CN107488649A (en) | A kind of fusion protein of Cpf1 and p300 Core domains, corresponding DNA target are to activation system and application | |
| WO2022159758A1 (en) | Novel engineered and chimeric nucleases | |
| KR102151064B1 (en) | Gene editing composition comprising sgRNAs with matched 5' nucleotide and gene editing method using the same | |
| CN114144519B (en) | Single base substitution protein and composition containing the same | |
| WO2021183850A1 (en) | Compositions and methods for modifying a target nucleic acid | |
| KR102679001B1 (en) | Compositions and methods for use of engineered base editing fusion protein | |
| WO2022147157A1 (en) | Novel nucleic acid-guided nucleases | |
| CN116529368A (en) | Type I-C CRISPR systems and methods of use from Neisseria lactis | |
| WO2025039972A1 (en) | Tls-based gene editing systems | |
| US20250059568A1 (en) | Class ii, type v crispr systems | |
| WO2024173573A1 (en) | Crispr-transposon systems and components | |
| WO2025085787A1 (en) | Engineered components of crispr and crispr-associated transposons systems | |
| HK1240617A1 (en) | Genome editing using campylobacter jejuni crispr/cas system-derived rgen | |
| HK1240617B (en) | Genome editing using campylobacter jejuni crispr/cas system-derived rgen |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| A201 | Request for examination | ||
| PA0109 | Patent application | St.27 status event code:A-0-1-A10-A12-nap-PA0109 | |
| PA0201 | Request for examination | St.27 status event code:A-1-2-D10-D11-exm-PA0201 | |
| PG1501 | Laying open of application | St.27 status event code:A-1-1-Q10-Q12-nap-PG1501 | |
| D13-X000 | Search requested | St.27 status event code:A-1-2-D10-D13-srh-X000 | |
| D14-X000 | Search report completed | St.27 status event code:A-1-2-D10-D14-srh-X000 | |
| E902 | Notification of reason for refusal | ||
| PE0902 | Notice of grounds for rejection | St.27 status event code:A-1-2-D10-D21-exm-PE0902 | |
| T11-X000 | Administrative time limit extension requested | St.27 status event code:U-3-3-T10-T11-oth-X000 | |
| T11-X000 | Administrative time limit extension requested | St.27 status event code:U-3-3-T10-T11-oth-X000 | |
| E13-X000 | Pre-grant limitation requested | St.27 status event code:A-2-3-E10-E13-lim-X000 | |
| P11-X000 | Amendment of application requested | St.27 status event code:A-2-2-P10-P11-nap-X000 | |
| P13-X000 | Application amended | St.27 status event code:A-2-2-P10-P13-nap-X000 | |
| E902 | Notification of reason for refusal | ||
| PE0902 | Notice of grounds for rejection | St.27 status event code:A-1-2-D10-D21-exm-PE0902 | |
| P11-X000 | Amendment of application requested | St.27 status event code:A-2-2-P10-P11-nap-X000 | |
| P13-X000 | Application amended | St.27 status event code:A-2-2-P10-P13-nap-X000 | |
| E701 | Decision to grant or registration of patent right | ||
| PE0701 | Decision of registration | St.27 status event code:A-1-2-D10-D22-exm-PE0701 | |
| GRNT | Written decision to grant | ||
| PR0701 | Registration of establishment | St.27 status event code:A-2-4-F10-F11-exm-PR0701 | |
| PR1002 | Payment of registration fee | St.27 status event code:A-2-2-U10-U11-oth-PR1002 Fee payment year number:1 | |
| PG1601 | Publication of registration | St.27 status event code:A-4-4-Q10-Q13-nap-PG1601 | |
| PR1001 | Payment of annual fee | St.27 status event code:A-4-4-U10-U11-oth-PR1001 Fee payment year number:4 | |
| PR1001 | Payment of annual fee | St.27 status event code:A-4-4-U10-U11-oth-PR1001 Fee payment year number:5 | |
| PR1001 | Payment of annual fee | St.27 status event code:A-4-4-U10-U11-oth-PR1001 Fee payment year number:6 |